状態空間モデルをstanでやりたかった①
注)本記事は途中で詰まったので結局モデル立てただけで結果は出てません。
状態空間モデルの勉強をしたものの、手を動かしていないので練習。
データ
kaggleのrossmann-storeコンペ。
カラム名の意味とかはここにまとまっている。
https://adtech.cyberagent.io/techblog/archives/259
アプローチ
練習なので、いったんトレンドと曜日効果のみで考える。
また、これはパネルデータなので、簡易化のためいったんある1店舗について考える。
②で、店舗タイプで縛った階層ベイズをやってみる予定。
モデル
以下のモデルで考える。
トレンド項
2階差分のトレンド項で考える。
これは、以下の式を変形することで求まる。これは、「当期 - 1期前の差分」と、「1期前 - 2期前の差分」は似ていることから以下のように表すことができるため。
似ているが微小に違う があるため、trendは滑らかに変化するという状態を表すことができている。
状態空間モデル
状態方程式
下記のような状態方程式を仮定する。
観測方程式
曜日成分
曜日は7日間の合計が0となる周期で表現する。
また、上式の を用いて以下のような表現で祝日効果、祝前日効果を付与することもできる。
・・・祝日効果:祝日は直近の日曜日と類似しているという仮定。
・・・祝日前効果:祝日前日は直近の金土曜日と類似しているという仮定。
ただしこのとき、 、
これらをまとめて、祝日要素も入れた曜日効果として、
※ はt日目が月〜金の祝日のとき1、それ以外で0。
はt日目が祝日でない月〜金かつ翌日が祝日のとき1、それ以外で0となる。
柔軟に曜日の効果を取り入れられる反面、パラメータが多いため時間がかかったり収束がしづらいという欠点がある。今回は簡略化のため1つ目の曜日7日間の周期効果のみ使用する。
プロモーション成分
以下のように定義する。もうちょっと細かく考えると、プロモーション効果は減衰していきそうなのでその点を踏まえたモデルにするとより良さそうだが、いったん定数で考える。
閉店日の影響
データは以下のようになっている(見づらいので一部期間のみ)。
df_sales = read_csv('./rossmann-store-sales/train.csv') %>% mutate(Store = as.factor(Store)) df_sales %>% filter(as.numeric(Store) ==1) %>% filter(Date < '2013-04-01') %>% ggplot(aes(Date, Sales, colour = Store)) + geom_line() + scale_x_date(labels = date_format("%Y/%m/%d"))
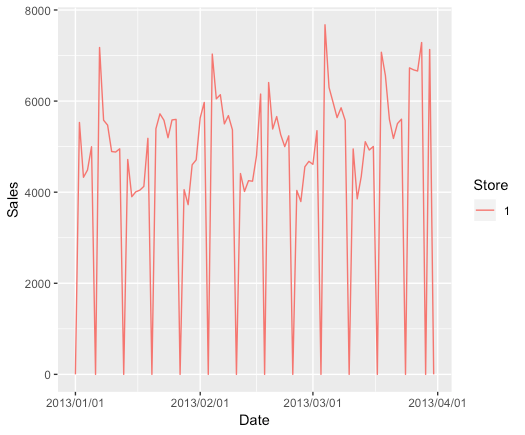
0となっている部分は閉店日。そのため、閉店日では0になるようする。
また、0部分を除くと以下のようになる。
df_sales %>% filter(as.numeric(Store) ==1) %>% #filter(Sales>0) %>% filter(Date < '2013-04-01') %>% ggplot(aes(Date, Sales, colour = Store)) + geom_line() + scale_x_date(labels = date_format("%Y/%m/%d"))
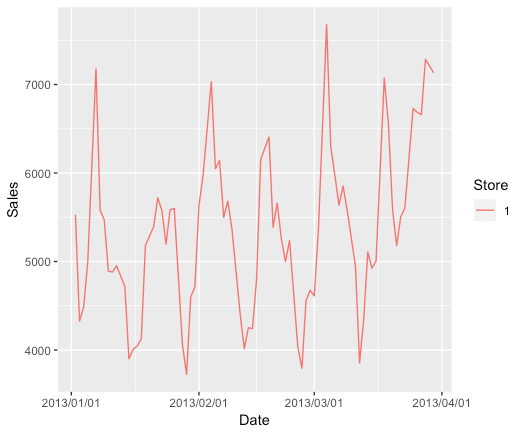
時系列では1時点前の値を参照することが多いが、今回1時点前が閉店日(Sales = 0) の場合は不適切となる。上図を見る限り、直近の開店日を参照しても問題がなさそうなのでそうすることにする。
以上を踏まえ、下記のような観測方程式を仮定する。
tが閉店日の場合、
それ以外の場合、
stanコード
data {
int<lower=1> N;
real SALES[N];
real<lower=0, upper=1> PROMO_FLG[N];
real<lower=0, upper=1> OPEN_FLG[N];
}
parameters {
real<lower=0, upper=10000> trend[N];
real dow[N];
real<lower=0, upper=100> sigma_trend;
real<lower=0, upper=100> sigma_dow;
real<lower=0, upper=100> sigma_sales;
}
transformed parameters {
real promo_effect[N];
real c_promo;
//プロモーション効果
for (i in 1:N) {
promo_effect[i] = c_promo * PROMO_FLG[i];
}
}
model {
// 状態方程式
for (i in 3:N) {
trend[i] ~ normal(2*trend[i-1] - trend[i-2], sigma_trend);
}
// 曜日効果
for (i in 7:N) {
dow[i] ~ normal(-dow[i-1] -dow[i-2] -dow[i-3] -dow[i-4] -dow[i-5] -dow[i-6], sigma_dow);
}
// 観測方程式
for (i in 3:N) {
if (OPEN_FLG[i] == 0){ // 閉店日
SALES[i] ~ normal(0,0); //0
} else {
SALES[i] ~ normal(trend[i] + dow[i] + promo_effect[i], sigma_sales);
}
}
}
generated quantities{
real sales_mu_all[N+3]; //3期先まで予測
real sales_pred[N+3];
real mu_all[N+3];
for (i in 1:N){ //実測部分まで
sales_mu_all[i] = trend[i] + dow[i] + promo_effect[i];
sales_pred[i] = normal_rng(sales_mu_all[i], sigma_sales);
}
for (i in N+1:N+3){ //予測部分
sales_mu_all[i] = normal_rng(sales_mu_all[i-1], sigma_sales); //状態
sales_pred[i] = normal_rng(sales_mu_all[i], sigma_sales); // 観測.閉店日の対応はしていないので注意
}
}
結果
Chain 1: Rejecting initial value: Chain 1: Error evaluating the log probability at the initial value. Chain 1: Exception: normal_lpdf: Location parameter is nan, but must be finite! (in 'model71f5bfe6db5_state_space_model' at line 46)
Chain 1: Chain 1: Initialization between (-2, 2) failed after 100 attempts. Chain 1: Try specifying initial values, reducing ranges of constrained values, or reparameterizing the model. [1] "Error in sampler$call_sampler(args_listi) : Initialization failed."
動かねーw
初期値が問題っぽいので、パラメータに制限をかけるべきなんだろうけど皆目検討つかない。てか無情報のdow[1~6], trend[1,2]あたりがなんとなく原因な気がする。
ちなみに
以下の簡単なモデルでは回った。
参考
RStanで『予測にいかす統計モデリングの基本』の売上データの分析をする - StatModeling Memorandum

予測にいかす統計モデリングの基本―ベイズ統計入門から応用まで (KS理工学専門書)
- 作者:樋口 知之
- 発売日: 2011/04/07
- メディア: 単行本
ベイズ推定において、ある変数間を縛りたい場合に置く仮定
表題通り、ある変数間を縛りたい場合に置く仮定について。
例えば、Z = B / Aのとき、AとBを個々にベイズ推定してZを求めるとする。
このとき、AにBがある程度連動する、つまり相関する場合は多変量正規分布を用いて取れる値を縛ると良い。
逆にいえば、A, Bが連動しているにも関わらず縛らない場合、つまりA, Bが自由に動いて組み合わされるのでZが真の範囲よりも非常に広い範囲で取られる可能性が高くなる。
こういったように、変数間でなにかしら共通の要因があるような変数同士の連動する振る舞いを仮定に入れたいときは 多変量正規分布 を用いるとよい。
多変量正規分布は、(上記の例で言えば)AとBが従う同時分布となっている。
パラメータとしては、平均ベクトルと分散共分散行列が必要となる。この分散共分散行列において、変数間の相関を定義することができる。
参考
状態空間モデルで何を状態方程式とし、何を観測方程式とするか
疑問
割と色々な書籍、例えばアヒル本で
観測値Y[t]を要素の和に分解するのが定石 (p.235)
とあるが、場合によるのでは?てかそもそも状態方程式/観測方程式どっちに何書くかの切り分けってなんだ?という疑問が湧いた。

StanとRでベイズ統計モデリング (Wonderful R)
- 作者:健太郎, 松浦
- 発売日: 2016/10/25
- メディア: 単行本
一般的によく書かれている簡易式は以下。
ここからもわかるように、状態方程式には 時刻とともに真の状態が確率的に変化する事象 を示し、観測方程式は 観測時に、真の状態に対して観測ノイズが加わる 。
つまり、時間変動するものを「状態方程式(状態項)」、その状態に時間不変のものが観測時に加わったものが「観測方程式」と考えられる。
例
1階差分TrendとEvengの有無(イベントフラグ)を用いた状態空間モデルを考える。今回、Eventの効果がタイミングによって不変か変化するかによる差を以下で示す。
例1:Eventの効果が時不変
以下のような構造の状態空間モデルを考える。
状態方程式
真の状態として、Trendの時間変動を考える。
観測方程式
観測するにあたって、真の状態に対して、イベント効果(時不変)と観測誤差が加わる。
例2:Eventの効果が時変
以下のような構造の状態空間モデルを考える。
状態方程式
真の状態として、Trendとイベント効果の時間変動を考える。
イベント効果の時間変動とは、イベントが起きたときの効果はタイミングによって異なることを意味する。
観測方程式
観測するにあたって、真の状態に対して、イベントの効果と観測誤差が加わる。
余談
まぁ状態方程式はなにか、観測方程式はなにか、ということは本質的にはどうでもいいので「状態空間モデルで何を状態方程式とし、何を観測方程式とするか」という議論自体にもあまり意味はないですがモデルを考える際の参考にはなりそう。
そういう状態/観測の切り分けなく書くと例1,2はそれぞれ以下のようになる(状態方程式を観測方程式に代入したものとイコール)。
ちなみに、今回は例えば「店舗売上を目的変数としたときに、近くでイベントがあったか」といったようなイベント効果を「あった/なかった」の「1/0」としている。その場合、前述のように時変のβは「近くでイベントがあったときの効果は日によって変わる」ことを示している。
これを例えば、「売上を目的変数としたとき、2018/01/01にCMを打った日以降か以前か」といった「1/0」とした場合、時変のβは「2018/01/01に打ったCMの、他の日への効果」、いわゆる 残存効果 を示すことになる。
Rでメモリを解放したい
デカイオブジェクトを何個も作る場合、メモリが足りなくなる。それゆえに、使わなくなったら削除してメモリを空けたい。
ただ、Rではどうやらメモリを食っているオブジェクトを削除するだけではメモリが空かないみたい(Rに限った話なのかは不明)。
以下は現在の使用メモリ量をみる。
15GB使っている模様。
>top -l 1 -s 0 | grep PhysMem PhysMem: 15G used (3186M wired), 17G unused.
なんかデカイオブジェクト作る。どうやら4.5GBなようだ。
tmp = matrix(rnorm(6e5*1000),6e5,1000) print(object.size(tmp), units = "auto") # => 4.5 Gb
再度、現在の使用メモリ量を見る。
>top -l 1 -s 0 | grep PhysMem PhysMem: 24G used (3192M wired), 8209M unused.
よくわからんが、先程作成したオブジェクトの4.5GB分ではなく、9GB分増えてる。想定より多いのはなんでかよくわからんが、とりあえず増えてるからこのまま進める。
先程作成した4.5GBのオブジェクトを削除する。
rm(tmp);
>top -l 1 -s 0 | grep PhysMem PhysMem: 24G used (3192M wired), 8209M unused.
オブジェクトを削除しても使用メモリ容量は変わってない。
ガーベージコレクションをしたら消えるみたい。おそらく、オブジェクト自体は削除されたけど、メモリが確保されたままなので強制解放をするってことなんかな。
ちなみに、2回連続でgc()しているのは以下を参考にしているからです(ただ1回だけでも消えてるような。。。)。
gc(reset = TRUE) gc(reset = TRUE) # gc(reset = TRUE) # used (Mb) gc trigger (Mb) limit (Mb) max used (Mb) # Ncells 1315929 70.3 2022381 108.1 NA 1315929 70.3 # Vcells 80286010 612.6 1806746712 13784.4 32768 80286010 612.6
>top -l 1 -s 0 | grep PhysMem PhysMem: 11G used (3173M wired), 21G unused.
はじめに書いた、巨大オブジェクト作成前の使用容量15GBより使用容量減ってるんですけど!tmpオブジェクト以外でも無駄に確保してたメモリも解放されたのか?
念の為、tmpオブジェクトを削除せずガーベージコレクションしたものと、その後にtmpオブジェクトを削除してからガーベージコレクションしたものの差分をみる。
tmpオブジェクトを削除せずガーベージコレクションすると15GB使用。
tmp = matrix(rnorm(6e5*1000),6e5,1000) gc(reset = TRUE);gc(reset = TRUE);
>top -l 1 -s 0 | grep PhysMem PhysMem: 15G used (3162M wired), 17G unused.
その後、tmpオブジェクトを削除してからガーベージコレクションすると11GB使用で、差分4GBなのでtmpオブジェクト分がおそらく浮いた模様。
rm(tmp); gc(reset = TRUE);gc(reset = TRUE);
>top -l 1 -s 0 | grep PhysMem PhysMem: 11G used (3163M wired), 21G unused.
参考
brmsを使ってみる
brmsというStanのラッパーパッケージで遊ぶ。
概要
例えば、rstanを使う場合はStanコードを別ファイルの.stanに記述してそれを呼び出す形でbayes推定をおこなう。一方、brmsを用いるとStanコードをわざわざ書かなくてもbrmsパッケージの関数を用いればbayes推定ができる。正確には、関数を介して内部的にStanコードを走らせているらしい。そのため、brmsを用いて書いたbayes modelが内部的に持っているStanコードはどうなっているか知りたい場合はそのコードを出力することも可能。
また、指定するための事前分布が豊富に存在するので、例えばStanで記述するのが面倒なゼロ過剰ポアソン分布なども簡単に使えるらしい。
ちなみに、brmsははBayesian Regression Models using Stanの略。
今回、brmsの練習のために「StanとRでベイズ統計モデル」のコードをいくつかbrmsで解く。

StanとRでベイズ統計モデリング (Wonderful R)
- 作者:健太郎, 松浦
- 発売日: 2016/10/25
- メディア: 単行本
線形回帰
以下のデータに対して重回帰をする。
library(tidyverse) library(tidylog) library(rstan) library(brms) rstan_options(auto_write = TRUE) options(mc.cores = parallel::detectCores()) df = read_csv('./chap05/input/data-attendance-1.txt') # # A tibble: 50 x 3 # A Score Y # <dbl> <dbl> <dbl> # 1 0 69 0.286 # 2 1 145 0.196 # 3 0 125 0.261 # 4 1 86 0.109 # 5 1 158 0.23 # 6 0 133 0.35 # 7 0 111 0.33 # 8 1 147 0.194 # 9 0 146 0.413 # 10 0 145 0.36 # # … with 40 more rows
OLS推定
まずはlmで以下をOLS推定。
lm_out = df %>% lm(Y ~ A + Score, data = .) summary(lm_out) # Call: # lm(formula = Y ~ A + Score, data = .) # # Residuals: # Min 1Q Median 3Q Max # -0.104746 -0.030294 -0.004062 0.026596 0.116984 # # Coefficients: # Estimate Std. Error t value Pr(>|t|) # (Intercept) 0.1233719 0.0323877 3.809 0.000404 *** # A -0.1437849 0.0145441 -9.886 4.63e-13 *** # Score 0.0016243 0.0002558 6.350 7.93e-08 *** # --- # Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 # # Residual standard error: 0.05038 on 47 degrees of freedom # Multiple R-squared: 0.7438, Adjusted R-squared: 0.7329 # F-statistic: 68.21 on 2 and 47 DF, p-value: 1.268e-14
結果は となる。
ベイズ推定
次に、brmを使ってベイズ推定。
ちなみに、オプションにsave_modelでmodel作成時に裏で走っているStanコードを.stanで保存できる。また、fileで作成したmodelを.rdsで保存できる。これはreadRDS関数で読み込める。
brm_out = df %>% brm(Y ~ A + Score, family="gaussian", data = ., save_model = "test_code", # test_code.stanという名前でstanファイルを保存 file = "test_model") # test_model.rdsという名前でmodelを保存 summary(brm_out) # Family: gaussian # Links: mu = identity; sigma = identity # Formula: Y ~ A + Score # Data: . (Number of observations: 50) # Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1; # total post-warmup samples = 4000 # # Population-Level Effects: # Estimate Est.Error l-95% CI u-95% CI Eff.Sample Rhat # Intercept 0.12 0.03 0.06 0.19 3988 1.00 # A -0.14 0.01 -0.17 -0.11 2029 1.00 # Score 0.00 0.00 0.00 0.00 4109 1.00 # # Family Specific Parameters: # Estimate Est.Error l-95% CI u-95% CI Eff.Sample Rhat # sigma 0.05 0.01 0.04 0.06 2018 1.00 # # Samples were drawn using sampling(NUTS). For each parameter, Eff.Sample # is a crude measure of effective sample size, and Rhat is the potential # scale reduction factor on split chains (at convergence, Rhat = 1).
結果は で、(下三桁以下がわからないが)OLSと同様になった。
Stanのsamplingの仕方は以下のようになっている(デフォルト)。
Samples: 4 chains, each with iter = 2000; warmup = 1000; thin = 1; total post-warmup samples = 4000
任意のものにしたい場合はオプションに指定する。
df %>% brm(Y ~ A + Score, family="gaussian", data = ., iter = 10, warmup = 10, seed = 1234, chain = 2)
今回familyにgaussianを使ったため、パラメータとしてmuとsigmaが生成される。それらのリンク関数は以下のように、identityとなる。
Links: mu = identity; sigma = identity
また、get_prior関数に作成したいmodelを入れると、そのmodelを使った場合に自動で作成される事前分布が表示される。
事前分布として、Intercept, sigmaともにstudent_t(3, 0, 10)が設定される。書いてないb(各回帰係数)は無情報事前分布が設定される。Intercept, sigmaが無情報じゃなくて弱情報分布なのは謎。sigmaはこのスライドみたいな理由かな?
df %>% get_prior(Y ~ A + Score, family="gaussian", data = .) # prior class coef group resp dpar nlpar bound # 1 b # 2 b A # 3 b Score # 4 student_t(3, 0, 10) Intercept # 5 student_t(3, 0, 10) sigma
任意の事前分布を入れたい場合はオプションにpriorを入れる。指定方法は何種類かある。
df %>% brm(Y ~ A + Score, family="gaussian", data = ., prior = c(prior_string("normal(0, 10)", class = "b"), # 文字列入れる指定方法 prior_string("normal(0, 10)", class = "b", coef = "A"), # 文字列入れる指定方法 prior(student_t(3, 19, 10), class = Intercept), # 直接入れる指定方法 prior_(~student_t(3, 0, 10), class = ~sigma) # ~と入れる指定方法 ) )
make_stancode関数にmodelを記載したらStanコードを吐く。
df %>% make_stancode(Y ~ A + Score, family="gaussian", data = ., prior = c(prior_string("normal(0, 10)", class = "b"), # 文字列入れる指定方法 prior_string("normal(0, 10)", class = "b", coef = "A"), # 文字列入れる指定方法 prior(student_t(3, 19, 10), class = Intercept), # 直接入れる指定方法 prior_(~student_t(3, 0, 10), class = ~sigma) # ~と入れる指定方法 ) )
また、コンパイル後でも内部的に走ったStanのコードも見ることができる。
こんな書き方しねー、って感じだけど基本的にはいい感じに早い書き方をしてくれる(らしい)。
brm_out$model`
# // generated with brms 2.8.0
# functions {
# }
# data {
# int<lower=1> N; // number of observations
# vector[N] Y; // response variable
# int<lower=1> K; // number of population-level effects
# matrix[N, K] X; // population-level design matrix
# int prior_only; // should the likelihood be ignored?
# }
# transformed data {
# int Kc = K - 1;
# matrix[N, K - 1] Xc; // centered version of X
# vector[K - 1] means_X; // column means of X before centering
# for (i in 2:K) {
# means_X[i - 1] = mean(X[, i]);
# Xc[, i - 1] = X[, i] - means_X[i - 1];
# }
# }
# parameters {
# vector[Kc] b; // population-level effects
# real temp_Intercept; // temporary intercept
# real<lower=0> sigma; // residual SD
# }
# transformed parameters {
# }
# model {
# vector[N] mu = temp_Intercept + Xc * b;
# // priors including all constants
# target += student_t_lpdf(temp_Intercept | 3, 0, 10);
# target += student_t_lpdf(sigma | 3, 0, 10)
# - 1 * student_t_lccdf(0 | 3, 0, 10);
# // likelihood including all constants
# if (!prior_only) {
# target += normal_lpdf(Y | mu, sigma);
# }
# }
# generated quantities {
# // actual population-level intercept
# real b_Intercept = temp_Intercept - dot_product(means_X, b);
# }
plot関数を用いると結果が可視化できる。他にも限界効果や交互作用を見るmarginal_effectsなどもある。
plot(brm_out) pp_check(brm_out)


ある程度はbrms内でできるが細かい可視化は、前回の記事で紹介したようなパッケージが使えるのでそちらに投げると良い。
launch_shiny(brm_out)
階層ベイズ
以下のようなモデルとそこから生成されるデータを考える。このモデルは推定量は会社毎に異なるが、業界が同じだとある程度似るモデル。
id X KID GID a b Y_sim 1 1 10 1 1 285.9306 9.499843 403.9857 2 2 28 2 2 336.9215 5.253227 535.2640 3 3 14 3 1 288.4397 15.351148 491.0800 4 4 31 4 2 306.2665 12.613492 639.5556 5 5 33 1 1 285.9306 9.499843 624.5689 6 6 1 2 2 336.9215 5.253227 324.4447
例えば以下の式において、 は
という固定効果と
に分解できる。
階層ベイズモデルを実装する:lme4とbrmsパッケージを用いたマルチレベルモデルの基礎
このような場合、brmsのformula式では ( X | 会社ID) という指定の仕方で表現することができる(おおもとで言えば、混合線形モデル用のパッケージlme4の書き方)。
そのため、今回のモデルでは以下のような書き方となる?
(1 + X | KID)に対して、1 + Xの1は切片、XはXに対する係数となり、それらに対してKIDグループ化(KIDで効果が異なる)、
(1 + X | GID)に対して、GIDでグループ化、ということを表している・・・らしい。
例えば切片部分(1)で考えるなら、 となり、業界が同じなら
が共通なので似た値になるということが実現されている。そのため、
KID GID どちらともで指定すると2階階層となる。
formula = Y_sim ~ 1 + X + (1 + X | KID) + (1 + X | GID)
また、全体的なコードとしては以下のようになる?事前分布として、[tex: X{会社} ~ Normal(X{業界共通}, \sigma)] とかそういう構造は下記のpriorの指定であってるのだろうか・・・?見たままだとNormal(0, 10)なのでなってない気がするが。。。このあたりはもうちょっとちゃんと調べます。
model = brm(Y_sim ~ 1 + X + (1 + X | KID) + (1 + X | GID), data = data, prior = c(prior_string("normal(0,10)", class = "sd", coef = "X", group = "KID"), prior_string("normal(0,10)", class = "sd", coef = "Intercept", group = "KID") ), chains = 4, iter = 1000, warmup = 500) summary(model)
Family: gaussian
Links: mu = identity; sigma = identity
Formula: Y_sim ~ 1 + X + (1 + X | KID) + (1 + X | GID)
Data: d (Number of observations: 40)
Samples: 4 chains, each with iter = 1000; warmup = 500; thin = 1;
total post-warmup samples = 2000
Group-Level Effects:
~GID (Number of levels: 2)
Estimate Est.Error l-95% CI u-95% CI Eff.Sample Rhat
sd(Intercept) 70.08 71.79 1.49 276.74 1060 1.00
sd(X) 10.13 10.92 0.21 42.22 261 1.02
cor(Intercept,X) -0.06 0.61 -0.96 0.94 1063 1.00
~KID (Number of levels: 4)
Estimate Est.Error l-95% CI u-95% CI Eff.Sample Rhat
sd(Intercept) 49.69 43.94 7.12 158.23 665 1.01
sd(X) 8.84 6.36 2.84 24.23 461 1.01
cor(Intercept,X) -0.43 0.49 -0.98 0.74 717 1.01
Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Eff.Sample Rhat
Intercept 311.99 60.61 191.35 449.71 861 1.00
X 8.30 4.46 -1.26 16.56 769 1.00
Family Specific Parameters:
Estimate Est.Error l-95% CI u-95% CI Eff.Sample Rhat
sigma 25.53 3.39 19.78 33.23 1305 1.00
Samples were drawn using sampling(NUTS). For each parameter, Eff.Sample
is a crude measure of effective sample size, and Rhat is the potential
scale reduction factor on split chains (at convergence, Rhat = 1).
警告メッセージ:
There were 169 divergent transitions after warmup. Increasing adapt_delta above 0.8 may help.
See http://mc-stan.org/misc/warnings.html#divergent-transitions-after-warmup
plot(model)


参考
https://cran.r-project.org/web/packages/brms/brms.pdf
Bayesian regression models using Stan in R | mages' blog
階層ベイズモデルを実装する:lme4とbrmsパッケージを用いたマルチレベルモデルの基礎
StanのMCMC結果&パラメータ結果を可視化する② tidybayes
Stanの結果を可視化する。
今回は tidybayesについて。
前回はbayesplot shinystanパッケージだった。
tidybayesでパラメータのサンプリング結果を可視化する
今回は以下の記事と公式を参考にします。
tidybayesパッケージで推定結果の整然化 - 竹林由武のブログ
Tidy Data and Geoms for Bayesian Models • tidybayes
tidybayesは、tidyなdplyrなどのtidyverse系で加工がしやすいイケてる感じでサンプリング結果を出してくれるパッケージ、らしい。
ちなみに前回書いたようにMCMC自体に対してサクっと見る関数は入ってないし、そこはもとからサポートする気はないみたい。
基本操作
まず、大本のfitの結果は以下。
> fit Inference for Stan model: model. 4 chains, each with iter=1000; warmup=500; thin=1; post-warmup draws per chain=500, total post-warmup draws=2000. mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat mu 8.18 0.31 5.59 -2.36 4.71 8.05 11.31 19.90 333 1.01 tau 6.79 0.29 5.58 0.25 2.61 5.48 9.55 21.50 369 1.01 eta[1] 0.40 0.02 0.89 -1.47 -0.19 0.43 0.99 2.10 2141 1.00 eta[2] 0.00 0.02 0.86 -1.70 -0.58 -0.02 0.59 1.65 2053 1.00 eta[3] -0.25 0.02 0.91 -1.97 -0.87 -0.25 0.32 1.59 1727 1.00 eta[4] -0.06 0.02 0.85 -1.74 -0.60 -0.05 0.49 1.64 1606 1.00 eta[5] -0.39 0.02 0.88 -2.10 -1.00 -0.39 0.19 1.36 1950 1.00 eta[6] -0.19 0.02 0.89 -1.96 -0.78 -0.21 0.42 1.49 1725 1.00 eta[7] 0.33 0.03 0.93 -1.65 -0.25 0.34 0.95 2.10 1313 1.00 eta[8] 0.04 0.02 0.99 -1.90 -0.63 0.04 0.71 1.98 1832 1.00 theta[1] 11.74 0.30 8.70 -1.79 6.16 10.36 15.69 33.96 836 1.00 theta[2] 7.90 0.13 6.24 -3.65 3.96 7.66 11.93 21.12 2253 1.00 theta[3] 5.74 0.20 7.77 -12.32 1.59 6.20 10.28 20.47 1454 1.00 theta[4] 7.55 0.17 6.52 -6.57 3.53 7.79 11.52 20.82 1412 1.00 theta[5] 4.97 0.15 6.42 -8.25 1.14 5.29 9.20 16.75 1833 1.00 theta[6] 6.31 0.16 6.87 -8.99 2.37 6.72 10.54 19.63 1953 1.00 theta[7] 10.70 0.17 6.77 -1.16 6.22 10.00 14.70 25.04 1520 1.00 theta[8] 8.23 0.20 8.08 -8.22 3.59 8.11 12.26 26.35 1620 1.00 lp__ -39.50 0.11 2.60 -45.19 -41.07 -39.25 -37.63 -35.18 571 1.01
くそ見づらい。tidybayesはspread_draws()に渡すとtidyにしてくれる。
以下の結果を見ればわかるが、どのchainのどのiterationなのかどのdrawなのか、どのr(listのindex)かという情報込で各パラメータが横持ちで全サンプリング結果をtibbleで取ってきてくれる。
下記ではeta[r]とtheta[r]はrが1~8まであるので各行でrに合わせて変わっている(r = 1はeta[1], theta[1])けど、tauとかはrがないため全て同じ値になっている。
fit_tidy = fit %>% spread_draws(tau, eta[r], theta[r], lp__) fit_tidy %>% head(10) # A tibble: 10 x 8 # Groups: r [8] .chain .iteration .draw tau r eta theta lp__ <int> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> 1 1 1 1 10.2 1 1.24 11.7 -38.4 2 1 1 1 10.2 2 0.845 7.66 -38.4 3 1 1 1 10.2 3 -0.889 -10.0 -38.4 4 1 1 1 10.2 4 1.07 9.90 -38.4 5 1 1 1 10.2 5 -0.900 -10.1 -38.4 6 1 1 1 10.2 6 -0.422 -5.27 -38.4 7 1 1 1 10.2 7 1.76 17.0 -38.4 8 1 1 1 10.2 8 1.00 9.26 -38.4 9 1 2 2 1.05 1 -0.843 8.26 -39.8 10 1 2 2 1.05 2 -0.661 8.45 -39.8
また、gather_draws()でパラメータを縦持ち化することもできる。
fit %>% gather_draws(c(tau, eta, theta, lp__)[r]) # A tibble: 32,000 x 6 # Groups: r, .variable [16] .chain .iteration .draw r .variable .value <int> <int> <int> <int> <chr> <dbl> 1 1 1 1 1 eta 1.26 2 1 1 1 2 eta -1.72 3 1 1 1 3 eta -1.02 4 1 1 1 4 eta -0.829 5 1 1 1 5 eta -0.242 6 1 1 1 6 eta -0.384 7 1 1 1 7 eta 0.895 8 1 1 1 8 eta -0.402 9 1 2 2 1 eta 0.186 10 1 2 2 2 eta -0.772 # … with 31,990 more rows
このspread_draws() gather_draws()を通した後のtidyなデータに対して、なにかを行うのがtidybayesの基本的な操作らしい。
事後分布
geom_eyeh()またはgeom_halfeyeh()で事後分布を求めることができる。前者だと上下両方に山ができ、後者だと片側のみの山。
どちらも、点推定値と確信区間を引数のpoint_intervalで指定した基準(中心値や平均値など)で出すことができる。
また、引数.widthで確信区間が何%確信区間かの指定ができる(デフォルトだと66,95%確信区間)。
このpoint_intervalオプションはtidybayes::point_interval()関数が内部的に呼ばれているので指定方法はpoint_interval関数のページを見れば良さそう。
例えば、point_intervalにはmedian_hdciやmean_qi、mode_qiなどを渡す。また、引数.widthはmedian_hdci関数などの引数と同じ。
point_interval function | R Documentation
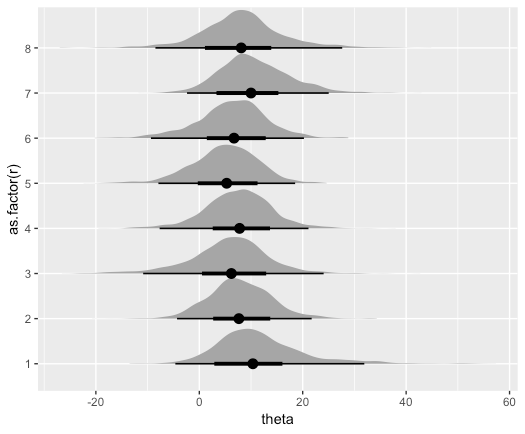
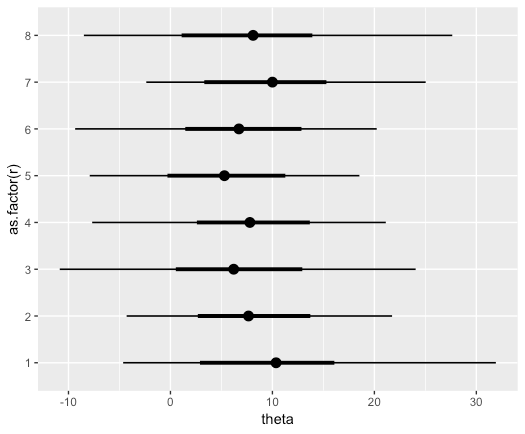
以下では、中心値に基づいた点推定値と、太線での66%確信区間、線での96%確信区間を伴った事後分布(片側山)となっている。
fit_tidy %>% ggplot(aes(theta, as.factor(r))) + geom_eyeh(point_interval=median_hdci,.width=c(.66,0.96))
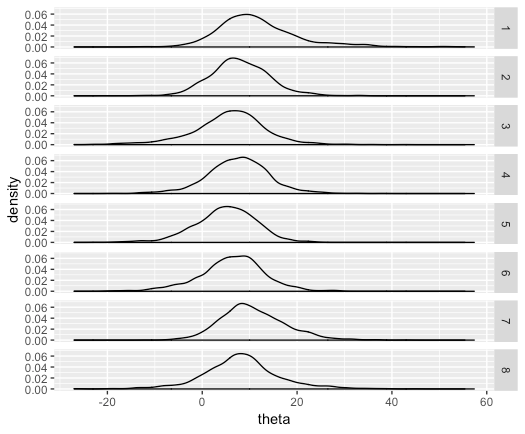
これもdplyr形式でやると以下のようになる(点推定値と確信区間はなし)。
fit_tidy %>% ggplot(aes(x=theta)) + geom_density() + facet_grid(r ~ .)
なお、stat_pointintervalh()を使った場合は密度関数の山がなしで点推定値と確信区間のみ表示される。
fit_tidy %>% ggplot(aes(theta, as.factor(r))) + stat_pointintervalh(point_interval=median_hdci,.width=c(.66,0.96))
統計量(point_interval関数系)
先程ふれたpoint_interval()関数系を使うと、値として得ることができる。point_interval()関数自体を使ってもいいし、median_qiのように、[median | mean | mode]_[qi | hdi | hdci] の組み合わせで直接指定することも可能。
- qi : quantile interval(パーセンタイル/分位点)
- hdi : Highest Density Interval (最高密度区間)
- hdci : High Dimensional Confidence Interval(?)
fit_tidy %>% median_hdci() # point_intervalを使うと以下になる #fit_tidy %>% #point_interval(.point = median, # .interval = hdci) # A tibble: 8 x 16 r tau tau.lower tau.upper eta eta.lower eta.upper theta theta.lower theta.upper <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> 1 1 5.48 0.000270 17.9 0.433 -1.56 1.95 10.4 -4.02 30.1 2 2 5.48 0.000270 17.9 -0.0207 -1.63 1.70 7.66 -4.30 20.1 3 3 5.48 0.000270 17.9 -0.251 -1.97 1.60 6.20 -11.3 21.1 4 4 5.48 0.000270 17.9 -0.0514 -1.72 1.66 7.79 -7.05 19.9 5 5 5.48 0.000270 17.9 -0.391 -1.94 1.48 5.29 -8.33 16.7 6 6 5.48 0.000270 17.9 -0.206 -1.89 1.54 6.72 -7.64 20.2 7 7 5.48 0.000270 17.9 0.338 -1.43 2.20 10.00 -1.35 24.8 8 8 5.48 0.000270 17.9 0.0396 -1.81 2.04 8.11 -9.24 25.2 # … with 6 more variables: lp__ <dbl>, lp__.lower <dbl>, lp__.upper <dbl>, .width <dbl>, # .point <chr>, .interval <chr>
ちなみにlower, upperを除いたmedian部分はただのdplyr処理だと以下で出せる。
fit_tidy %>% group_by(r) %>% summarize(tau = median(tau), eta = median(eta), theta = median(theta))
MCMC系
MCMCの収束診断などはbayesplotとか使えばいいからこのパッケージではフォローしないという公式の見解。ただ、bayesplotのための橋渡しとしてunspread_draws() ungather_draws()がある。これは、名前通りでspread_draws()やungather_draws()を行ってtidy型に加工したデータをもとのarray型に戻す動きをする。
これの何が嬉しいかというと、例えばspread_draws()でtidyにしたデータを加工した後にunspread_draws()でもとのarrayに戻し、それをbayesplotに渡してそっちで収束診断などをする、といった使用法ができる。
おまけ
bayesplotで同じようなことやりたい場合は以下を参考にするとよさそう。
Introduction to bayesplot (ppc_ series) - nora_goes_far
参考
StanのMCMC結果&パラメータ結果を可視化する① bayesplotとShinyStan
背景
最近rstan経由でStanを使ってる。rstanを用いた結果(収束診断とか事後確率分布とか)はそのままのデータでは可視化をするのが面倒。
可視化するのに便利なパッケージはないか調べてみると、ggmcmc とか bayesplot とか shinystan とか tidybayes とか色々ある模様。
友人に使い分けを聞いたりした感じだと、
- MCMCがうまくいってるかとか見るなら
bayesplotかshinystan - パラメータの事後分布(サンプリング結果)は
tidybayes
という使い分けがいいそうな。 とりあえずそれぞれで試してみる。
準備
データは以下のサイトと同様のものを使用する。
Introduction to bayesplot (mcmc_ series)
bayesplotでMCMC結果を見る
内容も上記サイトをトレースする。
概要
bayesplotではrstanの標準関数より便利な形でggplotオブジェクトとして結果を返してくれる。
bayesplotパッケージでは、大きく二つの関数系に分かれているとのこと - 1. mcmc xxx(収束や事後分布を確認するための関数群) - 2. ppc xxx(事後予測チェックを行うのに便利な関数群)
パラメータ推定が収束しているか
 の確認
の確認
rhat(fit)で書くパラメータの 結果を出力できる。
rhat(fit) # => mu tau eta[1] eta[2] eta[3] eta[4] eta[5] eta[6] eta[7] 1.0112444 1.0086249 1.0002212 1.0000860 1.0018156 0.9996789 1.0001951 0.9985509 1.0004175 eta[8] theta[1] theta[2] theta[3] theta[4] theta[5] theta[6] theta[7] theta[8] 1.0006213 1.0041166 0.9996360 1.0007257 1.0001193 1.0005308 1.0009002 1.0019892 1.0022765 lp__ 1.0126542
このデータをmcmc_rhat_hist()に渡すといい感じにrhatを出力してくれる。
は一般的に1.1以下なら良いため、その基準に基づいて凡例が色分けされている。
mcmc_rhat_hist(rhat(fit))

traceplotの確認
一度stanの結果をarrayにしてからmcmc_trace()にわたす。mcmc_combo()に渡した場合は事後分布と共に表示してくれる。
samples <- as.array(fit) mcmc_trace(samples, pars = "mu") ## n_warmupでwarmup区間指定可能 ## 事後分布と同時(パラメータ多いと重い) mcmc_combo(samples, pars = c("mu","tau"))

パラメータ推定が安定しているか
自己相関の確認
先ほどと同様に、配列型を関数に渡す。mcmc_acf()で折れ線、mcmc_acf_bar()で棒グラフとして、lagとの相関が(自己相関)が見れる。
library(patchwork) ## 折れ線表記 g1 = mcmc_acf(samples, pars = c("mu", "tau")) ## 棒グラフ表記 g2 = mcmc_acf_bar(samples, pars = c("mu", "tau")) g1 | g2

実行有効サンプルサイズ(ESS(n_eff))
0.1以下だと自己相関が高く、MCMCが上手くいってないとのこと(あんまよく理解してない
neff_ratio()でn_eff値を数値で、mcmc_neff()でn_eff値を図として確認できる。
# 値 neff_ratio(fit) # 図で出力 mcmc_neff_hist(neff_ratio(fit))

事後分布の確認
冒頭で書いたように、tidybayesの方が良さげだが一応。
確率密度
# chain合算 g3 = mcmc_dens(samples, pars = c("mu", "tau")) # chain毎 g4 = mcmc_dens_overlay(samples, pars = c("mu", "tau")) g3 / g4

まとめて1グラフに表示したい場合。上記のmcmc_dens()でもひとつといえばひとつなので好みか?
ただ、こっちのmcmc_areas()の場合は正規表現が使えるようになったり、信頼区間出たりする。
mcmc_areas(samples, c("mu", "tau"), prob = 0.95, prob_outer = 1, point_est = "mean") mcmc_areas(samples, regex_pars = "^eta\\[", prob = 0.95, prob_outer = 1, point_est = "mean")


mcmc_areas_data()で値として出力できる。
mcmc_areas_data(samples, regex_pars = "^eta\\[", prob = 0.95, prob_outer = 1, point_est = "mean") # A tibble: 16,534 x 5 parameter interval interval_width x density <fct> <chr> <dbl> <dbl> <dbl> 1 eta[1] inner 0.95 -1.47 0.0709 2 eta[1] inner 0.95 -1.46 0.0711 3 eta[1] inner 0.95 -1.46 0.0713 4 eta[1] inner 0.95 -1.45 0.0716 5 eta[1] inner 0.95 -1.45 0.0718 6 eta[1] inner 0.95 -1.45 0.0720 7 eta[1] inner 0.95 -1.44 0.0722 8 eta[1] inner 0.95 -1.44 0.0723 9 eta[1] inner 0.95 -1.44 0.0725 10 eta[1] inner 0.95 -1.43 0.0727 # … with 16,524 more rows
ヒストグラム
chain合算
mcmc_hist(samples, pars = c("mu", "tau"))

chain毎
mcmc_hist_by_chain(samples, pars = c("mu", "tau"))

散布図
mcmc_scatter(samples, pars = c("mu", "tau"))

散布図とヒストグラムまとめて
mcmc_pairs(samples, pars = c("mu", "tau"), diag_fun = c("hist")) mcmc_pairs(samples, pars = c("mu", "tau"), diag_fun = c("dens"))


ShinyStanでMCMC結果を見る
ShinyStanは名前通りShinyで動いている。
要はbayesplotとかで出力していることを、まとめてShinyアプリ上で勝手にplotしてくれるという優れもの。
rstanの結果のほかに、rstanarmやbrmsのような、ベイズ系パッケージに対しても使えるらしい。
グラフ画像を使ってなにかしたり、より自分好みの画像を作るとかじゃない限りは基本的にShinyStanを使えばいい気がする。
以下のように、作成したモデルに`launch_shinystan()'関数を適用すると起動する。
library(shinystan) my_sso = launch_shinystan(fit)

メニューはそれぞれ
- DIAGNOSE:MCMCの収束診断
- ESTIMATE:パラメータの推定結果
- EXPLORE:グラフ
- MORE:その他(コードや単語解説など)
となっている。
少し表示内容が変わっているが内容の詳細は以下のP37あたりを参照するとよい。
tidybayes
長くなったのでtidybayesは次の記事で。
参考
Plotting for Bayesian Models • bayesplot