StanのMCMC結果&パラメータ結果を可視化する② tidybayes
Stanの結果を可視化する。
今回は tidybayesについて。
前回はbayesplot shinystanパッケージだった。
tidybayesでパラメータのサンプリング結果を可視化する
今回は以下の記事と公式を参考にします。
tidybayesパッケージで推定結果の整然化 - 竹林由武のブログ
Tidy Data and Geoms for Bayesian Models • tidybayes
tidybayesは、tidyなdplyrなどのtidyverse系で加工がしやすいイケてる感じでサンプリング結果を出してくれるパッケージ、らしい。
ちなみに前回書いたようにMCMC自体に対してサクっと見る関数は入ってないし、そこはもとからサポートする気はないみたい。
基本操作
まず、大本のfitの結果は以下。
> fit Inference for Stan model: model. 4 chains, each with iter=1000; warmup=500; thin=1; post-warmup draws per chain=500, total post-warmup draws=2000. mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat mu 8.18 0.31 5.59 -2.36 4.71 8.05 11.31 19.90 333 1.01 tau 6.79 0.29 5.58 0.25 2.61 5.48 9.55 21.50 369 1.01 eta[1] 0.40 0.02 0.89 -1.47 -0.19 0.43 0.99 2.10 2141 1.00 eta[2] 0.00 0.02 0.86 -1.70 -0.58 -0.02 0.59 1.65 2053 1.00 eta[3] -0.25 0.02 0.91 -1.97 -0.87 -0.25 0.32 1.59 1727 1.00 eta[4] -0.06 0.02 0.85 -1.74 -0.60 -0.05 0.49 1.64 1606 1.00 eta[5] -0.39 0.02 0.88 -2.10 -1.00 -0.39 0.19 1.36 1950 1.00 eta[6] -0.19 0.02 0.89 -1.96 -0.78 -0.21 0.42 1.49 1725 1.00 eta[7] 0.33 0.03 0.93 -1.65 -0.25 0.34 0.95 2.10 1313 1.00 eta[8] 0.04 0.02 0.99 -1.90 -0.63 0.04 0.71 1.98 1832 1.00 theta[1] 11.74 0.30 8.70 -1.79 6.16 10.36 15.69 33.96 836 1.00 theta[2] 7.90 0.13 6.24 -3.65 3.96 7.66 11.93 21.12 2253 1.00 theta[3] 5.74 0.20 7.77 -12.32 1.59 6.20 10.28 20.47 1454 1.00 theta[4] 7.55 0.17 6.52 -6.57 3.53 7.79 11.52 20.82 1412 1.00 theta[5] 4.97 0.15 6.42 -8.25 1.14 5.29 9.20 16.75 1833 1.00 theta[6] 6.31 0.16 6.87 -8.99 2.37 6.72 10.54 19.63 1953 1.00 theta[7] 10.70 0.17 6.77 -1.16 6.22 10.00 14.70 25.04 1520 1.00 theta[8] 8.23 0.20 8.08 -8.22 3.59 8.11 12.26 26.35 1620 1.00 lp__ -39.50 0.11 2.60 -45.19 -41.07 -39.25 -37.63 -35.18 571 1.01
くそ見づらい。tidybayesはspread_draws()に渡すとtidyにしてくれる。
以下の結果を見ればわかるが、どのchainのどのiterationなのかどのdrawなのか、どのr(listのindex)かという情報込で各パラメータが横持ちで全サンプリング結果をtibbleで取ってきてくれる。
下記ではeta[r]とtheta[r]はrが1~8まであるので各行でrに合わせて変わっている(r = 1はeta[1], theta[1])けど、tauとかはrがないため全て同じ値になっている。
fit_tidy = fit %>% spread_draws(tau, eta[r], theta[r], lp__) fit_tidy %>% head(10) # A tibble: 10 x 8 # Groups: r [8] .chain .iteration .draw tau r eta theta lp__ <int> <int> <int> <dbl> <int> <dbl> <dbl> <dbl> 1 1 1 1 10.2 1 1.24 11.7 -38.4 2 1 1 1 10.2 2 0.845 7.66 -38.4 3 1 1 1 10.2 3 -0.889 -10.0 -38.4 4 1 1 1 10.2 4 1.07 9.90 -38.4 5 1 1 1 10.2 5 -0.900 -10.1 -38.4 6 1 1 1 10.2 6 -0.422 -5.27 -38.4 7 1 1 1 10.2 7 1.76 17.0 -38.4 8 1 1 1 10.2 8 1.00 9.26 -38.4 9 1 2 2 1.05 1 -0.843 8.26 -39.8 10 1 2 2 1.05 2 -0.661 8.45 -39.8
また、gather_draws()でパラメータを縦持ち化することもできる。
fit %>% gather_draws(c(tau, eta, theta, lp__)[r]) # A tibble: 32,000 x 6 # Groups: r, .variable [16] .chain .iteration .draw r .variable .value <int> <int> <int> <int> <chr> <dbl> 1 1 1 1 1 eta 1.26 2 1 1 1 2 eta -1.72 3 1 1 1 3 eta -1.02 4 1 1 1 4 eta -0.829 5 1 1 1 5 eta -0.242 6 1 1 1 6 eta -0.384 7 1 1 1 7 eta 0.895 8 1 1 1 8 eta -0.402 9 1 2 2 1 eta 0.186 10 1 2 2 2 eta -0.772 # … with 31,990 more rows
このspread_draws() gather_draws()を通した後のtidyなデータに対して、なにかを行うのがtidybayesの基本的な操作らしい。
事後分布
geom_eyeh()またはgeom_halfeyeh()で事後分布を求めることができる。前者だと上下両方に山ができ、後者だと片側のみの山。
どちらも、点推定値と確信区間を引数のpoint_intervalで指定した基準(中心値や平均値など)で出すことができる。
また、引数.widthで確信区間が何%確信区間かの指定ができる(デフォルトだと66,95%確信区間)。
このpoint_intervalオプションはtidybayes::point_interval()関数が内部的に呼ばれているので指定方法はpoint_interval関数のページを見れば良さそう。
例えば、point_intervalにはmedian_hdciやmean_qi、mode_qiなどを渡す。また、引数.widthはmedian_hdci関数などの引数と同じ。
point_interval function | R Documentation
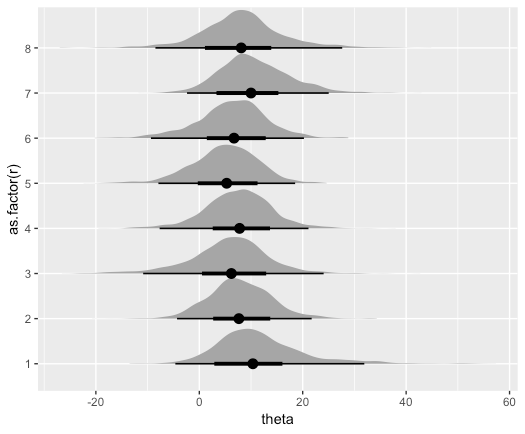
以下では、中心値に基づいた点推定値と、太線での66%確信区間、線での96%確信区間を伴った事後分布(片側山)となっている。
fit_tidy %>% ggplot(aes(theta, as.factor(r))) + geom_eyeh(point_interval=median_hdci,.width=c(.66,0.96))
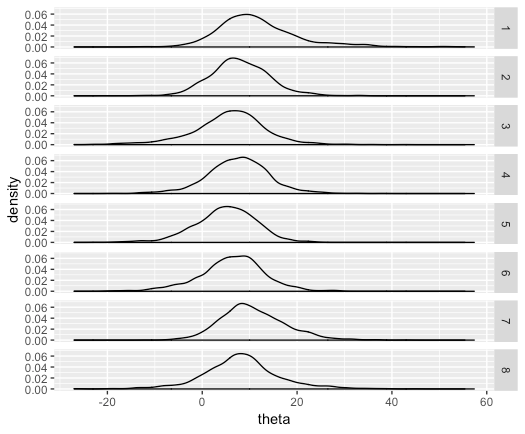
これもdplyr形式でやると以下のようになる(点推定値と確信区間はなし)。
fit_tidy %>% ggplot(aes(x=theta)) + geom_density() + facet_grid(r ~ .)
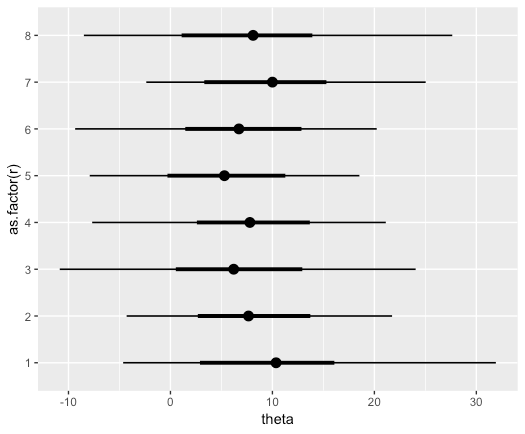
なお、stat_pointintervalh()を使った場合は密度関数の山がなしで点推定値と確信区間のみ表示される。
fit_tidy %>% ggplot(aes(theta, as.factor(r))) + stat_pointintervalh(point_interval=median_hdci,.width=c(.66,0.96))
統計量(point_interval関数系)
先程ふれたpoint_interval()関数系を使うと、値として得ることができる。point_interval()関数自体を使ってもいいし、median_qiのように、[median | mean | mode]_[qi | hdi | hdci] の組み合わせで直接指定することも可能。
- qi : quantile interval(パーセンタイル/分位点)
- hdi : Highest Density Interval (最高密度区間)
- hdci : High Dimensional Confidence Interval(?)
fit_tidy %>% median_hdci() # point_intervalを使うと以下になる #fit_tidy %>% #point_interval(.point = median, # .interval = hdci) # A tibble: 8 x 16 r tau tau.lower tau.upper eta eta.lower eta.upper theta theta.lower theta.upper <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> 1 1 5.48 0.000270 17.9 0.433 -1.56 1.95 10.4 -4.02 30.1 2 2 5.48 0.000270 17.9 -0.0207 -1.63 1.70 7.66 -4.30 20.1 3 3 5.48 0.000270 17.9 -0.251 -1.97 1.60 6.20 -11.3 21.1 4 4 5.48 0.000270 17.9 -0.0514 -1.72 1.66 7.79 -7.05 19.9 5 5 5.48 0.000270 17.9 -0.391 -1.94 1.48 5.29 -8.33 16.7 6 6 5.48 0.000270 17.9 -0.206 -1.89 1.54 6.72 -7.64 20.2 7 7 5.48 0.000270 17.9 0.338 -1.43 2.20 10.00 -1.35 24.8 8 8 5.48 0.000270 17.9 0.0396 -1.81 2.04 8.11 -9.24 25.2 # … with 6 more variables: lp__ <dbl>, lp__.lower <dbl>, lp__.upper <dbl>, .width <dbl>, # .point <chr>, .interval <chr>
ちなみにlower, upperを除いたmedian部分はただのdplyr処理だと以下で出せる。
fit_tidy %>% group_by(r) %>% summarize(tau = median(tau), eta = median(eta), theta = median(theta))
MCMC系
MCMCの収束診断などはbayesplotとか使えばいいからこのパッケージではフォローしないという公式の見解。ただ、bayesplotのための橋渡しとしてunspread_draws() ungather_draws()がある。これは、名前通りでspread_draws()やungather_draws()を行ってtidy型に加工したデータをもとのarray型に戻す動きをする。
これの何が嬉しいかというと、例えばspread_draws()でtidyにしたデータを加工した後にunspread_draws()でもとのarrayに戻し、それをbayesplotに渡してそっちで収束診断などをする、といった使用法ができる。
おまけ
bayesplotで同じようなことやりたい場合は以下を参考にするとよさそう。
Introduction to bayesplot (ppc_ series) - nora_goes_far