データサイエンス案件とアジャイル② DSに適したアジャイル詳細
データサイエンス案件とアジャイルについて。
前回に各手法についてざっと取り上げた。

- 作者:JonathanRasmusson,西村直人,角谷信太郎
- 発売日: 2017/07/14
- メディア: Kindle版
アジャイルにデータサイエンス案件をやる
アジャイルの哲学は 「不確実性をなくす」ために、考えられる全てのことを見える化・推定・認識のすり合わせをおこなうことで、できるだけその時点での不確実な要素の不確実性を除去して、クライアント・メンバーが満足するものを提供する こと。
そのため、基本的な流れは、アジャイルのところに書いた
具体的には、「インセプションデッキの作成」「ユーザーストーリーの作成」「工数概算見積もり」「計画づくり」といった準備のもと、毎スプリントで「各ストーリーの設計(深堀り)」「開発」「成果物発表」「フィードバック」という手順をおこなっていく。
のままで問題ないと思う。一方でデータサイエンス*1案件では、大枠で考えると「要件定義」に関する部分はソフトウェア開発でおこなうことがそのまま使えるが、「開発」および「成果物発表」はアレンジが必要になる。また「開発」にアレンジが加わるので連鎖的に「工数概算見積もり」「計画づくり」もアレンジが必要になる。
CRISP-DM/KDDとの対応
CRISP-DMの「ビジネス理解」はアジャイルの「インセプションデッキの作成」「ユーザーストーリーの作成」、「各ストーリーの設計(深堀り)」に相当する。
また、「データ理解」「データ準備」(= KDDの「データ獲得」「データ選択」「前処理」「変換」)「モデリング」が「開発」に、「解釈・評価」「共有・展開」が「成果物発表」「フィードバック」に相当する。

そのため、CRISP-DM/KDDを結合した以下のようなフローを考える。

開発
開発に関してソフトウェア開発では各ソフトの機能実装を優先順位に基づいてスプリントに入れていく。分析では機能は分析結果を出す以外はない。そのため、各スプリントではその「分析」をおこなうための各フローをおこなっていく。
より具体的には、下図の各フローの中身をユーザーストーリーベースで考えていくことになる*2。

また、この分析フローは結果を踏まえて何周も回す必要がある。何周もする前提なので、はじめから完璧なモデルを作成するのではなく、まずはベースラインとなる最低限動くモデルを作成し、成果物を発表しフィードバックをもらいその意見を反映しつつ徐々にリッチなモデルへと変えていく*3。
このように、フィードバックをもらうことを前提にしつつ周回をしていくので、1周 = 1スプリントと置くほうが都合が良いのでスプリント期間は1周にかかる期間に応じて考える。
なお、機械学習の場合は以前記事に書いたフローで考えた方がよさそう。
テスト駆動開発
開発する際に、やることは以下の4点となる。
- コードを書く
- テストをする
- リファクタリングをする
- レビューをする
データ分析では前述の分析フローを何周もするという性質上、 同じコードをある程度使いまわしていく。そのため、「テスト」「リファクタリング」「レビュー」が非常に重要となる。
例えば、前処理の「テスト」および「(テストに対する)レビュー」がテキトーだと、何周もした後に前処理がミスをしていたら今まで見ていたモデルの結果が全て意味のないものとなる。
また、同じコードに追加する、例えば前回前処理してできた変数をもとに追加の変数を作成したり、モデルの処理の一部を変えたり、といったことをする際にコードがきたないと追加をする際に非常にコストがかかるので「リファクタリング」および「(リファクタリングに対する)レビューが必要になる。
つまり、1開発につき「テスト」「リファクタリング」「レビュー」はセットでおこなっていく。
そのため、「テスト」「リファクタリング」を素早く回すためにアジャイルサムライで言及されているようにデータ分析でもテスト駆動開発をおこなった方がよい。
前処理・後処理部分でのテストは結局のところ「想定通りのデータとして処理できているか」の検証になるので、元データなどをもとにシンプルなダミーデータを作成してそのデータをもとにテストをおこなう。
ここでのやり方は現在考えているのでそのうち記事にまとめるが、Rの場合はassertを仕込むとテストがおこないやすいし、tidylogを使うと各tidyverse系の処理の過程がわかりやすくなるためこれらを使っておこなっていく。
モデル作成部分はテストのしようがないのでsummaryなどで係数の漏れやパラメータ指定の確認をする程度で、基本的にはレビュー任せか?
工数概算見積もり・計画づくり
前述のように「開発」は、分析結果を出す以外がないのでソフトウェア開発のように優先順位を基に決めることができず、分析フローに沿ったtodoを必要に応じて何周も回す必要がある。
つまり、データサイエンスでは何周するかで工数が変わる。そのため分析は工数が見積もりづらいので「工数概算見積もり」「計画づくり」をアレンジする必要がある。
具体的には分析フローを1周するときに各工程がどれくらいの工数が必要になるのかを見積もる。
そして「計画づくり」は、1周が作業量Xだとするとn周回したら作業量がnXになることをベースに考える。なお、一番初めの周は0から作るので作業量が多く、以降の周回は1周目のコードに手を加えていくことを考えると1周目よりも作業量が少ないことが多い。そのため前者を、後者を
とすると全体作業量は
となる。
ここから、「期限がxxxならn周回すことができる」「n周は最低限必要なので早くてxxxまでにはできあがる」という計画を作成する。

なお、実際は新たなデータを取得せずに既存データを変換して変数を増やしたりモデルを変えることが多く「データ理解」「データ獲得・選択」が発生しない周も多くなることが考えられるので1周目以外のはこれらを抜いた「前処理・モデリング」「解釈・評価」のみの労働量ではじめの計画づくりをした方が良いように思える*4。
また、以前紹介した Data Science and Agile: What works and what doesn’t work で言及されているように、分析では結果を踏まえてスコープや要求が変わりやすいが基本的にベースで労働量が増えるだけなので「残りの期限はxxxなので
から逆算するとこれくらいの追加は可能」という考え方ができる。
成果物発表
ソフトウェア開発では開発をしてできあがったものを動作させるだけでよいが、分析では開発結果(分析結果)だけではうまくいかないことが多い。成果物を発表する意図は「ちゃんと機能実装が完了しているか」「その機能がユーザーストーリーに即しているかのフィードバックを得る」ためにおこなう。
データサイエンスにおいて、なにか見せられる状態 =「ちゃんと機能実装が完了している」なので、この点は自然と終わっているので問題ない。
一方で「その機能がユーザーストーリーに即しているかのフィードバックを得る」は問題がある。
なぜなら、開発結果そのままだとフィードバックを得る際にコードから吐かれる結果をそのまま見せられても聞いている方は結果は見づらいし、そのままでは解釈もない。
そのため、データサイエンスでの「成果物」はコードの結果および解釈をなにかにまとめる必要があるので、「成果物発表」ではその点も加味して作業量を見積もる必要がある、
なお、前述のように成果物はフィードバックを得るために作るので成果物は「どういった分析をするか」「EDAの結果と解釈(モデルをどう作成するかの予定)」「分析結果とその解釈」となる。
つまり、これらの成果物を出し、
- 「どういった分析をするか」であれば分析の考慮漏れやよりよい分析アイデアがフィードバック
- 「EDAの結果と解釈(モデルをどう作成するかの予定)」であればEDAとして見るべき考慮漏れやEDAの結果の解釈やモデルアイデアがフィードバック
- 「分析結果とその解釈」であれば、分析からの知見や解釈、考慮すべき点のフィードバック
を得る。
まとめ
アジャイルはソフトウェア開発を前提に説明されているため、データサイエンスではそぐわない部分がある。しかし、アジャイルの哲学 「不確実性をなくす」ために、考えられる全てのことを見える化・推定・認識のすり合わせをおこなう*ことで、できるだけその時点での不確実な要素の不確実性を除去して、クライアント・メンバーが満足するものを提供する はデータサイエンスでも変わらないのでフレームワークの一部をデータサイエンス向けに変える必要がある。
具体的には「開発」の仕方がKDDベースの固定された内容に変わる。そして、開発はKDDを何周もする必要があるので「工数概算見積もり」「計画づくり」の考え方が変わる。
また、成果物の形態がソフトウェアと異なるため「成果物発表」を少し工夫する必要が生まれる。
まぁ正直やっていること自体はCRISP-DMとほぼ同じだが、「CRISP-DM」の「ビジネス理解」が抽象的だったが「インセプションデッキの作成」「ユーザーストーリーの作成」などより明確になった。更に、どうフローを回していくかといったことや期間の定め方が明確化した。
また、今回は変更がないので詳細は書いてないがその他アジャイルの進め方を模倣することでより不確実性が少なくなる。
このように、一部変更箇所はあるが、アジャイルを取り込むことによって、ソフトウェア開発と同様に不確実性をできるだけ排除したデータサイエンス案件ができるようになる。

その他
今回はあくまで「既存のソフトウェア開発でのアジャイルフレームワークをDSに落とすと際に変わる点」について記載した。
DSが共通部分含む各フレームワークでやることや、DSはどういうことを意識すればいいか、DS的にはどういう効力があるかといったような「DSにアジャイルを組み込むことの効果」は軽くしか書いていない。理由としては、共通部分を書くと「変わる点」がなにかわかりづらくなるため共通部分を書いていないが、そうなると「DSにアジャイルを組み込むことの効果」が書きづらいためあえて書いてない。
それらについては以下の記事が良かったのでご参照ください。
データサイエンス案件とアジャイル① 各既存手法まとめ
最近PL/PMすることが増えたので、チーム系の本(+マネジメント系の本)を読んだ。

チームが機能するとはどういうことか ― 「学習力」と「実行力」を高める実践アプローチ
- 作者:エイミー・C・エドモンドソン
- 発売日: 2014/09/05
- メディア: Kindle版

ハーバード・ビジネス・レビュー チームワーク論文ベスト10 チームワークの教科書
- 作者:ハーバード・ビジネス・レビュー編集部
- 発売日: 2019/03/07
- メディア: 単行本(ソフトカバー)
また、これらはチームに関して共通した運用方法・哲学なので DSプロジェクト自体の方法としていったんアジャイル系の本を読んだ。理由としては、近年色々なところでDSチームのアジャイル運用の話を聞くため。アジャイル自体はソフトウェア開発のプロジェクト運用フレームワークなので、そのまま運用はできないので、そもそもの哲学や使える場所を考える、という観点で読んだ。

- 作者:JonathanRasmusson,西村直人,角谷信太郎
- 発売日: 2017/07/14
- メディア: Kindle版
そのため、本記事では「DSでアジャイルにプロジェクトをすすめるにはどうしたらいいか」を考えるための前段として既存の使えそうなフレームワークを簡潔にまとめる。次回にそれらを踏まえた自分なりのアジャイルな分析案件の進め方をまとめる。
次回
分析フレームワーク
分析プロジェクトの既存フレームワークとしてCRISP-DMやKDDが存在する。
CRISP-DM
「ビジネス課題にはじまり、分s系により得られた利権をビジネス価値の向上に結びつけていく」という発想のフレームワーク。 「ビジネス理解」「データ理解」「データ準備」「モデリング」「評価」「共有・展開」の6つのステップに分け、それぞれをいったりきたりする。計画立案や、コードの整理などをおこなうときこのステップに分けて分割するときれいになることが多い。

KDD(Knowledge discovery in databases)
データ分析部分に特化して定義されたフレームワーク 「データ獲得」「データ選択」「前処理」「変換」「データマイニング」「解釈・評価」の6ステップに分かれる。
アジャイル
「不確実性をなくす」ために、考えられる全てのことを見える化・推定・認識のすり合わせをおこなうためのフロー。
基本的には「ソフトウェア開発」のための手法。
具体的には、「インセプションデッキの作成」「ユーザーストーリーの作成」「工数概算見積もり」「計画づくり」といった準備のもと、毎スプリントで「各ストーリーの設計(深堀り)」「開発」「テスト」「成果物発表」「フィードバック」という手順をおこなっていく。
この際、アジャイルチーム全員で各手順を一眼となって作成することで、チームメンバー内での認識の齟齬をなくすことができる。同時に、多くの手順ではクライアントとも一緒におこなうことで期待値コントロールも可能になる。
機能(todo)は1スプリント内で「設計→開発→実装」全てがおこなわれるとともに、クライアントからのフィードバックがセットでついてくる。そのため、期待値コントロールや要件とのズレの修正など柔軟性がある。
また、余談だが「共通の目的意識を持ち」「透明性がある」「公平性がある(それぞれが大切な役割を持つ)」「よりよい成果物を提供するために自己学習していく」というアジャイルの特徴は「チームが機能するとはどういうことか」など、チーム系の本で共通して出てくる良いチームの特徴となっている。つまり、不確実性をなくすだけではなくチーム運用も考えられたフレームワークとなっている。
DSにアジャイルが向いている部分向いてない部分
Data Science and Agile: What works and what doesn’t work
Data Science and Agile (Frameworks for Effectiveness)
ではアジャイル開発でDSに向いている部分向いてない部分を以下のようにまとめられている
向いている部分
- スプリント開始時の優先度付けと実行計画
- タスクを明確化する
- 各スプリント終了時のデモと振り返り
DSの作業でよくある間違いとして なんとなく分析をして本質と関係がない気になる箇所に時間を使ってしまう ということがよくある。
これを回避するために上述の3点をおこなうことで「目的をしっかり認識して」「それに必要な作業のみを洗い出し」「時間内に終わらす」ことができるし、認識のズレも極力なくすことができる*1。
向いてない部分と回避策
見積もりが困難
プロセスはある程度明確だが、各工程がどれくらいかかるかはデータによるため不明確。
これに対して、ストーリーポイントや工数を先に設定することでそれら 実験 をタイムボックス化することで回避する。
スコープや要求が変わりやすい
分析の過程でわかった内容をもとに、ステークホルダーから深堀りして欲しいという要求が生まれることが多い。
これに対して、常にステークホルダーと定期的な優先順位決めをおこない整理することで回避する。
各スプリントでデモをおこなう成果物が出しづらい
デモに必要な受け入れ基準、精度や改善インサイトはどれくらいでできるかスコープが切りづらい
これに対して、そもそもアジャイルはソフトウェア開発のフレームワークなので、DS作業では動くソフトウェアを出せないことを理解すること。代わりに、どういう実験をしたか、それを踏まえたネクストアクションはどうするか、といったような「そのスプリントでの実験と考察」をスプリント成果物として報告する。
スクラムは短絡的
クライアントの優先順位を優先したり期限を切ることで、根本が別だったりでより良くできることもスコープの範囲内での結果になってしまう
これに対して、例えばGoogleの20%ルールのようにイノベーションを起こすための時間を確保することで 効率的になりすぎる ことを回避できる。
DS向けアジャイル
前述の記事では、DSに適用した「タイムボック化されたスイテレーション(Time-boxed Iterations)」を用いたアジャイルを提言している。
Time-boxed Iterations
- 内容毎にフェーズ(タイムボックス)を分け、その中でいくつかのタスクを定める。各フェーズ内で満足する結果になったら次のフェーズへ、満足しなかったら再度そのフェーズを反復する。
- フェーズ毎にフィードバックをもらう(特に不明瞭さが多い初期ステージ
1. Feasibility Assessment
- ステークホルダーと要件(問題・要求・成果物・制約条件など)を定める
- ざっくりおこなうとどれくらいのパフォーマンス(精度)が出せそうか(ベースライン)
- 2~4week
2.Proof of Concept (POC)
- 最小実行可能なベースラインを作り、ステークホルダーに共有
- 性能要件を満たしているか、追加リソースが必要そうかを検証
- 4~8week
3. Deploy to Production
- 本番環境への移行
- 保守性・パフォーマンス・スケーラビリティ・保守性などのためにリファクタリングやドキュメント化
- 3-9ヶ月
4.Operational Maintenance
- 保守や新機能実装のメンテナンス

アジャイルにデータサイエンス案件をやる
基本的な流れは、アジャイルのところに書いた
具体的には、「インセプションデッキの作成」「ユーザーストーリーの作成」「工数概算見積もり」「計画づくり」といった準備のもと、毎スプリントで「各ストーリーの設計(深堀り)」「開発」「テスト」「成果物発表」「フィードバック」という手順をおこなっていく。
で問題ないと思う。
ただし、 成果物 がソフトウェア開発では機能なので、ここを分析結果となる。また、分析は工数が見積もりづらいので「工数概算見積もり」「計画づくり」に少し修正を加えることことが必要になるし、テストも「データ系のテストとは?」、成果物はなに?といったようにちょくちょくDS向けのやり方があるように思う。
これらを踏まえて、具体的になにをするか次回で記載する。
なお、内容は「アジャイルサムライ」をベースに記載する。
参考
*1:それぞれの詳細はリンク先を読んでください
Tidyevalでの関数型プログラミング俺俺メモ
プログラミングをするときにはDRYの法則と言われるように、同じような処理は関数で書いて使い回す方がメンテ効率や可読性が上がる。
特に、データサイエンス的な場合はコードの使い捨てがしやすいので煩雑になるので特に意識した方が良い。
一方で、Rのtidyverseは超便利だけど、関数を中心とした関数型プログラミング的な書き方、つまり変数を用いて色々変えつつやるときにNSEとかが絡んできて面倒。
過去にtidyverseとNSEについて書いたが、結局使うときにちゃんとできてないなぁといった感じなのでそれぞれでの書き方をまとめる。
なお、そのようなtidyevalという概念の詳細は過去記事に書いている。
要点として、tidyverse系は通常のSE表現(Standard Evaluation)ではなく、NSE表現(Non-Standard Evaluation)を使っているので表現式(expression)で書こう、という話。
全体的な話は公式ガイドを読むとよさげ
書き方系
!!
!!をQuosureに与えるとその箇所はquoteではなくなる。つまり、評価がされるようになる。なお、一部だけに適応したい場合は()で囲む
quo, enquo
quo, unquoはQuosureを作成する。また、!!と組み合わせることで、遅延評価が可能となる。例えば、
library(tidyverse) df = as_tibble(iris) df %>% filter((!!hoge) == 'versicolor') %>% ## hogeというquoteとして渡したあと、!!でquoteを一時的に外して評価される head()
と書くと、hogeは!!によって評価されるようになるのでhogeに何かを代入しておくと代入された項目が使える。
しかし、
hoge = Species df %>% filter((!!hoge) == 'versicolor') %>% ## hogeというquoteとして渡したあと、!!でquoteを一時的に外して評価される head()
とすると、Speciesというオブジェクトが代入時に評価されるが、Speciesはこの段階では定義されてないのでエラーとなる。また、ここを仮にhoge = 'Species'とした場合、文字列Speciesとして定義されているのでエラーにはならないがNSEでは以下のように、Species列ではなく文字列Species列となり、そんな列は存在しないので0行抽出される。
df %>% filter('Species' == 'versicolor') %>% ## hogeというquoteとして渡したあと、!!でquoteを一時的に外して評価される head() # A tibble: 0 x 5 # … with 5 variables: Sepal.Length <dbl>, Sepal.Width <dbl>, Petal.Length <dbl>, Petal.Width <dbl>, Species <fct>
このとき、前述のようにquoで遅延評価にしておくとfilter内で初めて評価がおこなわれ、そのときはdfでSpeciesは定義されているのでdfでの定義を用いてデータを呼び出すことになる。
# 変数hoge hoge = quo(Species) # Speciesを遅延評価 df %>% filter((!!hoge) == 'versicolor') %>% ## hogeというquoteとして渡したあと、!!でquoteを一時的に外して評価される head() ## A tibble: 6 x 5 # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # <dbl> <dbl> <dbl> <dbl> <fct> # 1 7 3.2 4.7 1.4 versicolor # 2 6.4 3.2 4.5 1.5 versicolor # 3 6.9 3.1 4.9 1.5 versicolor # 4 5.5 2.3 4 1.3 versicolor # 5 6.5 2.8 4.6 1.5 versicolor # 6 5.7 2.8 4.5 1.3 versicolor
他にも、enquoというものがあり、シンボル(変数とか引数とか)に対するquo。
厳密には、遅延評価なのは同じだが、quoは環境がlocal、unquoは環境がglobalとなる。
つまり、関数内で使用する場合、
quo()では実行されているその環境を用いるので、渡された引数をそのままexprに設定する。
enquo()では関数の呼び出し元の環境を用いるので、その呼び出し元の環境で引数を評価したものをexprに設定する。
そのため、関数の引数を使う場合はquoではなくunquoを使う。
なお、文字列で定義したい場合はsym で一度シンボル化する。
piyo = sym('Species') # シンボル化 # piyo = as.name('Species') # こっちでもよい hoge = enquo(piyo) # Speciesを遅延評価 df %>% filter((!!hoge) == 'versicolor') %>% ## hogeというquoteとして渡したあと、!!でquoteを一時的に外して評価される head() ## A tibble: 6 x 5 # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # <dbl> <dbl> <dbl> <dbl> <fct> # 1 7 3.2 4.7 1.4 versicolor # 2 6.4 3.2 4.5 1.5 versicolor # 3 6.9 3.1 4.9 1.5 versicolor # 4 5.5 2.3 4 1.3 versicolor # 5 6.5 2.8 4.6 1.5 versicolor # 6 5.7 2.8 4.5 1.3 versicolor
:=
mutateやfileterの左側の列名に!!を用いて展開した値を使用する場合は、右側とを=で繋ぐのではなく:=をセットで用いる必要がある。
tmp_hoge = as.name('hoge') # シンボル化 hoge = enquo(tmp_hoge) # Speciesを遅延評価 df %>% mutate((!!hoge) := Species) %>% ## hogeというquoteとして渡したあと、!!でquoteを一時的に外して評価される head() # # A tibble: 6 x 6 # Sepal.Length Sepal.Width Petal.Length Petal.Width Species hoge # <dbl> <dbl> <dbl> <dbl> <fct> <fct> # 1 5.1 3.5 1.4 0.2 setosa setosa # 2 4.9 3 1.4 0.2 setosa setosa # 3 4.7 3.2 1.3 0.2 setosa setosa # 4 4.6 3.1 1.5 0.2 setosa setosa # 5 5 3.6 1.4 0.2 setosa setosa # 6 5.4 3.9 1.7 0.4 setosa setosa # 同じ結果 hoge = quo(hoge) # Speciesを遅延評価 df %>% mutate((!!hoge) := Species) %>% ## hogeというquoteとして渡したあと、!!でquoteを一時的に外して評価される head() # # A tibble: 6 x 6 # Sepal.Length Sepal.Width Petal.Length Petal.Width Species hoge # <dbl> <dbl> <dbl> <dbl> <fct> <fct> # 1 5.1 3.5 1.4 0.2 setosa setosa # 2 4.9 3 1.4 0.2 setosa setosa # 3 4.7 3.2 1.3 0.2 setosa setosa # 4 4.6 3.1 1.5 0.2 setosa setosa # 5 5 3.6 1.4 0.2 setosa setosa # 6 5.4 3.9 1.7 0.4 setosa setosa
各関数での使い方サンプル
各関数で実際どう書くか羅列。
前述の公式ガイドにクックブックもある。
mutate
f_mutate = function(df, mutate_col, col1, col2) { mutate_col = enquo(mutate_col) col1 = enquo(col1) col2 = enquo(col2) df = df %>% mutate((!!mutate_col) := (!!col1) * (!!col2)) return(df) } f_mutate(df, col_by, Sepal.Length, Sepal.Width) # # A tibble: 150 x 6 # Sepal.Length Sepal.Width Petal.Length Petal.Width Species col_by # <dbl> <dbl> <dbl> <dbl> <fct> <dbl> # 1 5.1 3.5 1.4 0.2 setosa 17.8 # 2 4.9 3 1.4 0.2 setosa 14.7 # 3 4.7 3.2 1.3 0.2 setosa 15.0 # 4 4.6 3.1 1.5 0.2 setosa 14.3 # 5 5 3.6 1.4 0.2 setosa 18 # 6 5.4 3.9 1.7 0.4 setosa 21.1 # 7 4.6 3.4 1.4 0.3 setosa 15.6 # 8 5 3.4 1.5 0.2 setosa 17 # 9 4.4 2.9 1.4 0.2 setosa 12.8 # 10 4.9 3.1 1.5 0.1 setosa 15.2 # # … with 140 more rows
filter
f_filter = function(df, filtered_col, filtered_val) { filtered_col = enquo(filtered_col) df = df %>% filter((!!filtered_col) == filtered_val) return(df) } f_filter(df, Species, 'setosa') # # A tibble: 50 x 5 # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # <dbl> <dbl> <dbl> <dbl> <fct> # 1 5.1 3.5 1.4 0.2 setosa # 2 4.9 3 1.4 0.2 setosa # 3 4.7 3.2 1.3 0.2 setosa # 4 4.6 3.1 1.5 0.2 setosa # 5 5 3.6 1.4 0.2 setosa # 6 5.4 3.9 1.7 0.4 setosa # 7 4.6 3.4 1.4 0.3 setosa # 8 5 3.4 1.5 0.2 setosa # 9 4.4 2.9 1.4 0.2 setosa # 10 4.9 3.1 1.5 0.1 setosa
select
f_select = function(df, col1, col2) { col1 = enquo(col1) col2 = enquo(col2) df = df %>% select((!!col1), (!!col2)) return(df) } f_select(df, Sepal.Length, Sepal.Width) # # A tibble: 150 x 2 # Sepal.Length Sepal.Width # <dbl> <dbl> # 1 5.1 3.5 # 2 4.9 3 # 3 4.7 3.2 # 4 4.6 3.1 # 5 5 3.6 # 6 5.4 3.9 # 7 4.6 3.4 # 8 5 3.4 # 9 4.4 2.9 # 10 4.9 3.1 # # … with 140 more rows
group_by, summarise
f_summarise = function(df, group_col, summarise_col) { group_col = enquo(group_col) summarise_col = enquo(summarise_col) df = df %>% group_by(!!group_col) %>% summarise((!!summarise_col) := mean((!!summarise_col))) return(df) } f_summarise(df, Species, Sepal.Length) # # A tibble: 3 x 2 # Species Sepal.Length # * <fct> <dbl> # 1 setosa 5.01 # 2 versicolor 5.94 # 3 virginica 6.59
ggplot
f_plot = function(df, var_x) { var_x = enquo(var_x) g = df %>% ggplot(aes((!!var_x), Sepal.Width)) + geom_point() return(g) } f_plot(df, Sepal.Width)
おまけ
expr関数を使うと、今コードが最終的にどういう状態になっているか確認ができる。
mutate
f_mutate = function(df, mutate_col, col1, col2) { mutate_col = enquo(mutate_col) col1 = enquo(col1) col2 = enquo(col2) df = df %>% mutate((!!mutate_col) := (!!col1) * (!!col2)) return(df) } f_mutate(df, col_by, Sepal.Length, Sepal.Width) # f_mutateでなにが起きているか確認 expr_f_mutate = function(df, mutate_col, col1, col2) { mutate_col = enquo(mutate_col) col1 = enquo(col1) col2 = enquo(col2) expr_f = expr(df %>% mutate((!!mutate_col) := (!!col1) * (!!col2))) return(expr_f) } expr_f_mutate(df, col_by, Sepal.Length, Sepal.Width) # df %>% mutate(`:=`(~col_by, (~Sepal.Length) * ~Sepal.Width))
select
f_select = function(df, col1, col2) { col1 = enquo(col1) col2 = enquo(col2) df = df %>% select((!!col1), (!!col2)) return(df) } f_select(df, Sepal.Length, Sepal.Width) # # A tibble: 150 x 2 # Sepal.Length Sepal.Width # <dbl> <dbl> # 1 5.1 3.5 # 2 4.9 3 # 3 4.7 3.2 # 4 4.6 3.1 # 5 5 3.6 # 6 5.4 3.9 # 7 4.6 3.4 # 8 5 3.4 # 9 4.4 2.9 # 10 4.9 3.1 # # … with 140 more rows # f_selectでなにが起きているか確認 expr_f_select = function(df, col1, col2) { col1 = enquo(col1) col2 = enquo(col2) expr_df = expr(df %>% select((!!col1), (!!col2))) return(expr_df) } expr_f_select(df, Sepal.Length, Sepal.Width) # => df %>% select(~Sepal.Length, ~Sepal.Width)
参考
正規表現で文字列からほしい部分を抽出する
Rでstr_match_all(string, pattern)でstringに対してマッチしたpattern文字列を抽出する。
pattern部分は()を入れることでpattern中の一部分だけを取ることができる。
listの1要素目がマッチした部分の全体、2要素目以降が括弧でマッチさせた抽出される。
_allがないstr_matchの場合ははじめに一致したもののみlistで取得。
_allありはstr_matchの場合は一致したものがすべてMatrixで取得。
実例
model_hoge:fuga_piyo:fugaという文字列から、_と:に挟まれた部分hogeとpiyoを取り出したい。
patternとしては、_, _以外の文字列複数文字, : というpatternのうち、_以外の文字列複数文字を抽出したい。そのため、以下になる
library(tidyverse) str_i = 'model_hoge:fuga_piyo:fuga' # str_match_all str_matced_i_all = str_match_all(str_i, '_([^_])+:') # [[1]] # [,1] [,2] # [1,] "_hoge:fuga" "hoge" # [2,] "_piyo:fuga" "piyo" str_matced_i_all[[1]][1,2] # => hoge str_matced_i_all[[1]][2,2] # => piyo # str_match str_i_matced = str_match(str_i, "_([^_])+:fuga") # [,1] [,2] # [1,] "_hoge:fuga" "hoge" str_i_matced[2] # => hoge
正規表現部分解説
patternの'([^]+):'について。
[^]は[]内にある文字以外を指す。そのため、[^_]は_以外の文字列となり、[^_]+は_以外の文字複数個となる。
つまり、「'([^]+):'は_と:で囲まれた_以外の文字複数個部分」がマッチするうち、()内の_以外の文字複数個が抽出される。
今回わざわざ_以外の文字複数個としているのは、文字が_..._..._となっていて_が繰り返されている部分も一致するのでそうしないと_を含む箇所も抽出されてしまうため。
str_matced = str_match_all(str_i, "_(.+):fuga") # [[1]] # [,1] [,2] # [1,] "_hoge:fuga_piyo:fuga" "hoge:fuga_piyo"
参考
pythonで動的にオブジェクトを作成する
目的
pythonで動的にオブジェクトを変えたい。
ユースケース
乱数を変えたランダムサンプリングデータを複数個作りたいときや、絞り込みを変えたデータを作成したいときなど。
execでのやり方
execを使うと文字列でコードを書けるので利用する。
# ランダムサンプリング for i in range(4): exec(f'data_sample{i} = data.sample(n=100)') # => data_sample1, data_sample2, data_sample3, data_sample4ができあがる # 絞り込みを変えたデータを作成 j_list = ['hoge', 'fuga', 'piyo'] for j in j_list: exec(f'df_{j} = df.query("type == '{j}'")') # => df_hoge, df_fuga, df_piyoができあがる
dictを使ったやり方
execはまぁ直感的だからわかりやすい。ただし、変数名が明示的ではなかったり、スコープがよくわからんことになるので他の方法で実現できる方法の方がベスト。
# ランダムサンプリング d = dict() for i in range(3): d[i] = df.sample(n=100) # => d[0]などでアクセス可能 # 絞り込みを変えたデータを作成 d2 = dict() j_list = ['hoge', 'fuga', 'piyo'] for j in j_list: d2[j] = df.query("type == '{j}'") # => d2['hoge']などでアクセス可能
参考
git俺俺メモ
Git(Github)の最低限checkout add commit pushはしているがそれくらいしかわからんので、これどーしたらいいっけというのをメモ。
用語
working tree: ファイルの最新状態。addすることでstage(index)に遷移。
stage(index): ファイルのaddした状態。commitすることでlocal repositoryに遷移。
local repository(HEAD): 最新のcommit状態(直前におこなったcommit状態)。現在のlocal repositoryともいえる。
HEAD^: 1つ前のcommit状態
working tree → stage → local repositoryと遷移する。ここまでは自分の作業環境(local)の話で、ここからpushをすることでlocal repositoryを共有の作業場であるremote repositporyにあげることができる。
この記事の図がとてもわかりやすい。
Gitコマンド
addを取り消す
いくつかまとめてaddをしてしまったがcommitをすると全addがcommitされる。このとき、一部のみcommit対象にしたい場合はaddしたファイルを取り消す。
つまり、addでstagedになっている一部ファイルを最新のcommit状態(HEAD)に戻すのでgit reset HEAD file_name。
最新のcommit状態→何か修正→Addなので、修正部分が消えるように思えるが、git reset HEAD file_nameは正確にはgit reset HEAD --mixed file_nameなので、 インデックスの変更(addした内容)のみ を戻している。
なお、同じファイルにcommitせず複数回Addをしている場合はまとめて1回のAddと同じ意味なのでまとめてAdd自体は取り消される(修正内容は破棄されないが、stagedにはない状態)。
また、git reset --hard HEAD file_name だと、stage, working treeの変更 = commit以降の全変更が取り消されて最新のcommitした状態になる(=変更は破棄)。
なお、以下の記事ではいろいろな「もとに戻す」がまとめられている。
複数commitを1つにまとめたい
commitした後に細かいミスを見つけたため再度commitして修正する、、、みたいなことをすると無駄に2commitになってわかりづらい。
そういうときはcommit をsquachして1つにまとめる。
branchにoriginの情報を取り込む
branchにいるときはbranchを切ったとき段階での状態だが、その間にリモートリポジトリ(origin)が更新されると更新されたファイルはそのbranch上では最新版でなくなる。 fetch&merge?
作業途中で別branchの作業をする
あるbranckの作業中に別branch(または新規作業内容)が差込で入った。
一回commitしてからbranchを移動することもできるが、いい感じのcommit粒度じゃない場合commitをしたくない。
そのようなときは、stashをする。
git stash save 'メッセージ' でcommitしていないもの(addしてないもの含む)一時的に退避する。メッセージは省略可。
stash@{0}: WIP on test: xxxx みたいな感じで表示される。xxxはメッセージが表示される。メッセージを省略した場合、HEAD(直前のcommit)時のメッセージが表示される(※ちゃんとcommit以降のaddなどの変更部分も含めて退避される(。
git stash list でstash一覧が表示でき、git stash pop stash@{N} でN番目のstashを復元する。stash@{N}を省略すると最新のstashが適用される。
git stash show stash@{N} でN番目に変更したファイルの一覧を、git stash show -p stash@{N} でN番目に変更した差分一覧が表示できる。
これでいったん現branchでの作業途中を退避できるのでいったんcommitをしなくても別branchに移動することができる。
状態間の差分
branchで作業しているときに、他タスクが差込で入ったりしたときに「どこまでやったっけ?」となる。
また、「作業した記憶ないけどなんでこれ変更された扱いになってるんだ?」ということも個人的にはちょくちょくある(Rスクリプトで、commitしたあとに一瞬データを見るために出力コマンドだけ書いたりしたときによくある)。
基本的に、git diff [状態1] [状態2] [file name] で状態1と2の[file name]の差分を見ることができる。
また、git diff以降に指定するオプションなどでどの状態間を比較するかが変わるし、[状態]部分も省略によって変わる。
直前のAdd(staged)から現在の変更部分を見る
直前のAdd(staged)から現在の変更部分はgit diff [file path] で見られる。
最新のcommit(HEAD)から現在の変更部分を見る
最新のcommit = HEADから、現在とHEADの差分なので git diff HEAD [file path] で変更箇所が見れる。
なお、[file path]を指定しないとHEAD ~ 現在で変更があった全ファイルの変更点が全て確認できる。
変更があったファイルの確認はgit status
直前のAdd(staged)から最新のcommit(HEAD)の変更部分を見る
直前のAdd = stagedなので、git diff --staged [file path]で確認できる。
cachedも同じ意味なのでgit diff --cached [file path]でもよい。
直前のAdd(stage)から現在の差分は表示されない
最新のcommitとその前のcommitの差分を見る
基本形のgit diff [状態1] [状態2] [file name] における、状態1 = HEAD^、状態2=HEADとなるため git diff HEAD^ HEADで見ることができる。
直近のcommitメッセージを取り消す(push前)
タイピングミスや内容のミスをした場合など。
直前のcommitの場合はgit commit --amendで修正をする。
直前よりも前の場合、まずはgit log --oneline で直近の操作logを見ていつのcommitか調べる。
その後、git rebase -i HEAD~nでn番目までのcommit履歴のpickをeditにして上書き保存で閉じてからメッセージの修正をし、git commit --amendをする。
一部ファイルを管理対象外にする
基本的に決まってるファイルはgitignoreに追加するとして、データソースはgitにあげたくないときはいちいち追加するのが面倒なので、ignoreではなく都度データソース用フォルダ毎まとめて指定をしたいときなど。
git rm --cache file_name
フォルダの場合は-rをつける。
git rm --cached -r cache/
gitignoreは既に管理対象の場合は記述しても外れないらしい。
オプションなしでgit rm file_nameだとファイル自体が削除されるので注意。
Gitでのやりとり
commit粒度
どういう粒度でcommitをするか。
「xxxの修正と、yyy機能の追加と、リファクタリングしました!」みたいなcommitだとどのdiffがどれかよくわからん。
そのため、commitの粒度は意識する。
例えば、上記だと
- xxxの修正
- yyy機能の追加
- リファクタリング
の3commitに分けた方が良い。そうするとそれぞれのcommitのdiffを見ると「そのdiffは何をしたやつなのか」が明確にわかる。
なお、上記3commit粒度は例えば「xxxの修正」は「xxxの修正のために、データ処理の変更」「処理の変更に伴い影響がある部分を修正」のように更に細かく分けることができる場合もある。
このようなときは必要に応じて更にcommitを分けたほうが良い。
結局、「どういう粒度ならレビュー者(および未来の自分)がわかりやすいか?」ということを考えてcommitする必要がある。
commitメッセージ
そのcommitが「バグの修正」なのか「機能の追加」なのか「リファクタリング」なのか、メッセージ文章を読めばわかるかもしれないがパっと見で理解できると便利。
そのため、メッセージの接頭に
- バグの修正なら
Fix - 機能の追加なら
Add - リファクタリングなら
Refaxt
と付けるなどするとレビュー者の理解の助けになる。
以下の記事がよくまとまっている。
プルリクの書き方
- 変更内容の詳細(要約はcommit メッセージに書いている)
- どういう観点で見てほしいか
- 特に見て欲しい箇所
- 注意点
- プルリク作業想定時間
- 期限
また、分析系の業務の場合はデータそのものが無いと処理の理解がしづらいので、場合によっては読み込むデータをGoogle Driveなどにアップロードするとよりよいかも*1。
また本記事の趣旨とはそれるが、データは各処理をするたびにちゃんとコメントをしたり、headした結果を貼っているとレビュー者の負担が減るかも。まぁコードが煩雑になるというデメリットはあるが。。。
また、コミットメッセージは複数行書くことができる。
デファクトスタンダードとしては、1行目に50文字以内で要約、2行目を空白、3行目以降は詳細を書くのが基本なようだ。
なお、コミットメッセージはcommit -m 'hoge' -m '' -m 'hoge'のように複数回-mをすると複数行メッセージを書ける。また、 -mなしだとvimでの記載となる。
*1:データ容量がデカいからgitに上げたくないとかあると思うので、自分は基本的にgit上ではなくDriveにアップロードしている
BUSINESS DATA SCIENCE 6章 Controls
BUSINESS DATA SCIENCEの続き。
データなどは作者のgitにある。
最近日本語版も出たみたいです

- 作者:マット・タディ
- 発売日: 2020/07/22
- メディア: 単行本
この章はなにか
5章*1では実験環境下、つまりRCTの場合の因果効果を測る話だったが6章はそうでない場合、conditional ignorabilityを用いることで、効果を測りたいdのyへの共変量XでControlをおこなうことで、yへのdだけの効果を取り出す。つまり、共変量Xを用いることで擬似的にRCTと同様の環境にする。
5章・6章(の一部)の話は「効果検証入門」のときにもやっている
本書ではそこから一歩先に進んで、共変量Xを選ぶのは難しいので自動的にLassoでXを選択する方法を紹介している。また、全体的な平均処置効果Average Treatment Effect(ATE)だけではなくある要素毎での処置効果(例えば、性別毎での処置の違いや年収ごとなど)となるHeterogeneous Treatment Effect(HTE)の紹介もおこなっている。
Conditional Ignorability
Conditional Ignorability(以下CI)は以下の式で表される。
これは、共変量xが与えられたときに、効果を検証したい項目d(バイナリの場合は割当)はどのdの場合のyの同時分布とも独立している ことをいう。つまり、dの値は共変量xによってのみ決まり、yによっては決まらない。言い換えると、xが同じならdは同じ値になる。そのため、dとyのストレートな関係を得られるといえるし、RCTと同様になるためセレクションバイアスがかからないともいえる。
書き方を変えると、 および
となる。
なお、これは「効果検証入門」でConditional Independence Assumption(CIA)が書かれていたが(IgnorabilityとIndependenceの違いはあれど)CIAをd(Z)=1/0(バイナリ)ではなく一般化したもの?
CIA =
また、これらより強い条件としてStrongly Ignorable Treatment Assignment(強く無視できる割当条件) というものがあり、「どのx(Z)も観測値」+ 「どのiもどのdになる可能性もある」という条件が加わる模様。このあたりは各著者がどういう意図で書き分けてるのかは謎。おおもとのRubinはStrongly Ignorable Treatment Assignmentを置いて効果検証をしているっぽい。
実践
オレンジジュースデータを用いて、値段が売上に与える効果を考える。まずは単回帰をおこなう。
basefit <- lm(log(sales) ~ log(price), data=oj) coef(basefit) # (Intercept) log(price) # 10.423422 -1.601307
このときの-1.601307は、値段に対して価格の割当が一定でない、つまり共変量Xが含まれていない。仮に共変量がオレンジジュースのブランドのみだとすると、以下では共変量X=ブランドを加えることで値段の売上に対する割当がランダムとなる。ブランドをA,B,Cとして式で書くと、
となる。つまり、「ブランドさえ同じなら、例えば値段がいくらであろうと、それぞれでの売上の確率分布は同じ」だし、「ブランドさえ同じなら、売上がいくらであろうと値段の確率分布は同じ」ということ。逆に、「ブランド毎で、同じ値段のときの売上の確率分布が変わる」ともいえる。
そのため、ブランドを式に入れてControllすることで、「ブランドによって売上の確率分布が変わる」のを起きなくさせて、値段を通したブランドによる売上への効果を除去した、値段と売上のストレートな関係を観ることができる。
brandfit <- lm(log(sales) ~ brand + log(price), data=oj) coef(brandfit) # (Intercept) brandminute.maid brandtropicana log(price) # 10.8288216 0.8701747 1.5299428 -3.1386914
You can better contorol for confounder by modeling the treatment assignment process
LTE
CIAを明示的に回帰式に組み込むと以下のLinear Treatment Effect(LTE)で表すことができる。
①
②
dは処置、xはyに対するdの全共変量を指す。x'は yに対するdの共変量の一部(不完全な共変量)を指している。
ここで がCIAを指している。
つまり、2段目(②)の式 によって、x`に足りない全共変量xのdへの影響がνとして表される。
そして、そのx', dを使った1段目(②)の式 では不完全な共変量x'を使っているが、dはνを用いることで擬似的に全共変量xを含めた値となっていることからγは全共変量xでContorollしたyへの効果を示すことになる。
要するに、全共変量xを求めるのは難しいので、不完全な共変量x'だけで上手くdのyへの効果を抽出するのがLTEとなっている。
ただし、これは共変量xの次元が少ない(次元<<n)の場合を前提としているため高次元xのときは成り立たない。そのため、できる限り重要な共変量をxとして入れて低次元化する必要があるがどれを選んだらいいか試すのは非常に時間がかかるのでlassoとCVを用いることで解決させる。
LTE LassoRegression
LTEをLasso経由でうまく求める。
- d = E(d|x) = x`τ としてML(Lasso)で学習をおこない、実際のデータにfittingさせて
を求める。
をLassoで求める(
はモデルに必要なので除外さえないように罰則項を入れない)
このとき2.で推定されたが効果(treatment effect)になる。
より詳細に書くと、1.で求めた と真のdの差分は
となる。この
はLTEの式②よりx = x'であれば
となることから、
は共変量の一部であるx'で表せきれていない共変量xのdへの効果を捉えていると考えられる。
そのため、LTEの式① に式②
を代入すると、
となり、変形すると
となる。
とすると
になり、これとLTEの式①を見比べると、「効果をみたい変数d」の係数と「
= 共変量の一部であるx'で表せきれていない共変量xのdへの効果」の係数はどちらもγとなる。
2.では「でControllした
の係数γ」は、「
の効果」となる。「
の効果」は「 dの全共変量でコントロールしたときの効果」となることから、2.式より、dの全共変量でコントロールしたときの効果を係数γから求めることができる。
実践
以下の殺人率データを用いて、ケータイ電話の所持率の効果をみる。
sna <- factor(s, levels=c(NA,levels(s)), exclude=NULL) x = sparse.model.matrix( ~ t + phone*sna + .^2, data=controls)[,-1] # 共変量x' # 1.部分 treat <- cv.gamlr(x ,d, lmr=1e-3) # E[d|x] = x'τを作成 dhat <- drop( predict(treat, x, select="min") ) # treatを使ってdhatを求める # 2.部分 causal <- cv.gamlr(cBind(d,dhat,x),y,free=2,lmr=1e-3) # dhatのみpenaltyを与えない coef(causal, select="min")["d",] # 共変量で統制したdのyへの効果 # => 0.
Sample-Splitting and Orthogonal Machine Learning
LTE LassoではLassoを用いて効果を求めている。Lassoでは通常の方法では信頼区間を求めることができない。しかし、3章で紹介したような方法(※当ブログでは書いてない)でLassoに対してモデル選択後の係数信頼区間をパラメトリックブートストラップとサブサンプリングで求める方法を紹介している。
LTE Lassoでもその方法は可能だが、LTEモデルを用いたLassoに関してより簡易な方法として、データを2分割し、
- 片方のデータでLassoでモデル選択をおこなう
- そのモデルでOLSをおこなう
ことで、OLSが噛むためOLS部分で信頼区間を求めることができる。また、これをCross Validationでおこなうことで汎化的な値を求める。
具体的には、
- データをK分割する
- 1-1. k群のデータで、LassoなどのMLを用いて E[d | x]とE[y | x] *2のモデル選択をおこなう
- 1-2. k群以外のデータで1-1.のモデルを用いたd, y の予実差
を求める
1-3. 上記をk+1, k+2,...Kでもおこなう
を求め、
の信頼区間を求める
LTE LassoRegressionの以下
① d = E[d|x] = x`τ としてML(Lasso)で学習をおこない、実際のデータにfittingさせて を求める。
② = \hat{d}δ + dγ + x'β] をLassoで求める(
はモデルに必要なので除外さえないように罰則項を入れない)
と対応させて考えると、1-1. 1-2. は①に当てはまる。そのため、予実差は、x(x')だけでは表せきれていない共変量の効果
となる。
また、E[y | x] から作成したはx(x')だけでは表せきれていないy部分となる。
そのため、 と
の関係式の
の係数は2.
のように効果γとなる。
言い換えると、「yの共変量xで表せきれていない部分」と「dの共変量xで表せきれていない部分」の関係性となるのでそれは効果と等しくなる。
そしてこれはOLSなのでγの信頼区間も合わせて求めることができる、
実践
上記アルゴリズムは以下から読み込める
source("examples//orthoML.R") dreg <- function(x,d){ gamlr(x, d, standardize=FALSE, lmr=1e-5) } yreg <- function(x,d){ gamlr(x, d, family="binomial", standardize=FALSE, lmr=1e-5) } resids <- orthoPLTE( x=x[,-who], d=x[,who], y=y, dreg=dreg, yreg=yreg, nfold=5) # fold: 1 2 3 4 5 # gamma (se) = 0.238736 (0.0212952) 0.238736 + c(-2,2)*0.0212952 # 信頼区間2se # 0.1961456 0.2813264 2*pnorm(-0.238736/0.0212952) # p値 # => 3.609601e-29
Heterogeneous Treatment Effect
先程まで観ていた効果は、処置有無毎でのAverage Treatment Effect(ATE)となっている。このATEは、例えば男女混合での効果だが男性への効果/女性への効果、のようにあるグループに分けた効果をHeterogeneous Treatment Effect(HTE)と呼ぶ。
ビジネスにおいて、効果が高いHeterogeneousがどれかを把握することは重要なためHTEは非常に役に立つ。
これは、
となる。このx'は共変量+ Heterogeneousとなる。はbaseとなる効果で、交互作用
によって、共変量, Heterogeneousそれぞれの効果がベクトルγで現れる。そのため、Heterogeneousの効果は
で現れる。
なお、式からわかるようにHeterogeneous毎にサブサンプリングをおこなって通常のLTEをおこなうことでもHeterogeneousの効果を表すことはできる。
また、これより例えばA,Bテストの場合
となる。
実践
データは以下
まずは、Heterogeneousとしてみたい項目XのNAを無くす。
### linear HTE estimation (Controls chapter) library(gamlr) source("examples/naref.R") ## dealing with missingness # make NA the reference level for all categories X <- naref(X) # pull out the numeric variables xnum <- X[,sapply(X,class)%in%c("numeric","integer")] xnum[66:70,] colSums(is.na(xnum)) # flag missing xnumna <- apply(is.na(xnum), 2, as.numeric) xnumna[66:70,] # impute the missing values mzimpute <- function(v){ if(mean(v==0,na.rm=TRUE) > 0.5) impt <- 0 else impt <- mean(v, na.rm=TRUE) v[is.na(v)] <- impt return(v) } xnum <- apply(xnum, 2, mzimpute) xnum[66:70,] # replace/add the variables in new data frame for(v in colnames(xnum)){ X[,v] <- xnum[,v] X[,paste(v,"NA", sep=".")] <- xnumna[,v] } X[144:147,]
次に先程のみたい項目X(Heterogeneous)と共変量 P$numhhを結合してx'を作成する。
xhte <- sparse.model.matrix(~., data=cbind(numhh=P$numhh, X))[,-1] xhte[1:2,1:4] dim(xhte) # 6855 64 P %>% head() # person_id household_id doc_any_12m selected medicaid numhh # 1 1 100001 0 1 0 1 # 2 2 100002 0 1 1 1 # 5 5 100005 0 1 0 1 # 6 6 100006 1 1 0 1 # 8 8 102094 0 0 0 2 # 9 9 100009 1 0 0 1
次に、x'とdの交互作用項を作成。
dxhte <- P$selected*xhte colnames(dxhte) <- paste("d",colnames(xhte), sep=".") # 列名をd.x名とする htedesign <- cBind(xhte,d=P$selected,dxhte)
そして、x'と交互作用項をあわせたhtedesignを用いてLassoを行う。
# include the numhh controls and baseline treatment without penalty htefit <- gamlr(x=htedesign, y=P$doc_any_12m, free=c("numhh2","numhh3+","d")) # numhh2, numhh3, d列は罰則項なし gam <- coef(htefit)[-(1:(ncol(xhte)+1)), ]
このときgamにd.Xの交互作用項の係数をgamに格納している。これをいくつか見てみる。
round(sort(gam)[1:6],4) round(sort(gam, decreasing=TRUE)[1:6],4)
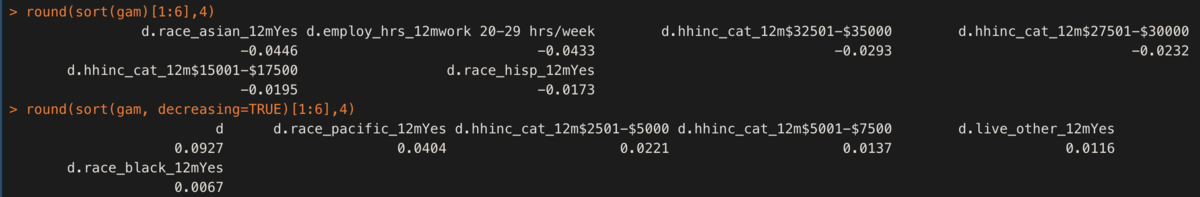
dとなっているものが今回効果をみたいもの(selected)のbase効果となる。ここから、例えばrace_pacific_12mYesであることでd+race_pacific_12mYes = 0.0927+0.0404 = 0.133つまり、13%増加する効果がある。
なお、ATEは [\hat{γ_0} + x'平均 \hat{γ_0} ]で計算できる。
gam["d"] + colMeans(xhte)%*%gam[grep("d.", names(gam))] # => 0.05616546
その他
今回でBUSINESS DATA SCIENCEの勉強は終わり。他にも自然言語処理とかの章もあるが使わないのでいったん保留。
全体的にビジネスという観点で必要なものをどうやって出すか、ということで実用的だった。
2点だめな点をあげると、Rの書き方が今風(tidyverse)ではないこと、sampledataが散在していること。
何のデータを使うか単位でRファイルがあり、そのファイルは色々な章で登場する。そのため、例えば1,5章でデータXを使う場合1,5章のコードが全てX.Rに記載されている。そして、5章は1章部分のコードの続きという立ち位置なので1章部分を読み込まないと5章部分だけでは動かない。
また、書籍内コードにread_csv部分の記載やどのRファイルを使うか書いてないにも関わらず、ファイル名がわかりづらい。つまり、毎回どのファイルか調べないといけないので糞めんどくさい。

