mutate_atで引数付き関数を使う
やりたいこと
mutateでは、関数を以下のように適用を適用することができる。
library(tidyverse) iris %>% group_by(Species) %>% mutate(Sepal.Length_lag2 = lag(Sepal.Length,2)) # Sepal.Length Sepal.Width Petal.Length Petal.Width Species Sepal.Length_lag2 # <dbl> <dbl> <dbl> <dbl> <fct> <dbl> # 1 5.1 3.5 1.4 0.2 setosa NA # 2 4.9 3 1.4 0.2 setosa NA # 3 4.7 3.2 1.3 0.2 setosa 5.1 # 4 4.6 3.1 1.5 0.2 setosa 4.9 # 5 5 3.6 1.4 0.2 setosa 4.7 # 6 5.4 3.9 1.7 0.4 setosa 4.6 # 7 4.6 3.4 1.4 0.3 setosa 5 # 8 5 3.4 1.5 0.2 setosa 5.4 # 9 4.4 2.9 1.4 0.2 setosa 4.6 # 10 4.9 3.1 1.5 0.1 setosa 5
また、mutate_atではvarsに指定した列に対して関数を適用できる。funsで作成したlist(fun_list)を関数部分に用いると指定した要素名を末尾に追加した列名を追加することができる。
iris %>% group_by(Species) %>% mutate_at(vars(Sepal.Length:Petal.Width), funs('lag' = lag)) # # Sepal.Length Sepal.Width Petal.Length Petal.Width Species Sepal.Length_lag Sepal.Width_lag Petal.Length_lag Petal.Width_lag # <dbl> <dbl> <dbl> <dbl> <fct> <dbl> <dbl> <dbl> <dbl> # 1 5.1 3.5 1.4 0.2 setosa NA NA NA NA # 2 4.9 3 1.4 0.2 setosa 5.1 3.5 1.4 0.2 # 3 4.7 3.2 1.3 0.2 setosa 4.9 3 1.4 0.2 # 4 4.6 3.1 1.5 0.2 setosa 4.7 3.2 1.3 0.2 # 5 5 3.6 1.4 0.2 setosa 4.6 3.1 1.5 0.2 # 6 5.4 3.9 1.7 0.4 setosa 5 3.6 1.4 0.2 # 7 4.6 3.4 1.4 0.3 setosa 5.4 3.9 1.7 0.4 # 8 5 3.4 1.5 0.2 setosa 4.6 3.4 1.4 0.3 # 9 4.4 2.9 1.4 0.2 setosa 5 3.4 1.5 0.2 # 10 4.9 3.1 1.5 0.1 setosa 4.4 2.9 1.4 0.2
しかし、このときfunsを使った関数で引数を使おうとしても上手く動かない(何故か全てNA)になる。
iris %>% group_by(Species) %>% mutate_at(vars(Sepal.Length:Petal.Width), funs('lag' = lag(2))) # Sepal.Length Sepal.Width Petal.Length Petal.Width Species Sepal.Length_lag Sepal.Width_lag Petal.Length_lag Petal.Width_lag # <dbl> <dbl> <dbl> <dbl> <fct> <dbl> <dbl> <dbl> <dbl> # 1 5.1 3.5 1.4 0.2 setosa NA NA NA NA # 2 4.9 3 1.4 0.2 setosa NA NA NA NA # 3 4.7 3.2 1.3 0.2 setosa NA NA NA NA # 4 4.6 3.1 1.5 0.2 setosa NA NA NA NA # 5 5 3.6 1.4 0.2 setosa NA NA NA NA # 6 5.4 3.9 1.7 0.4 setosa NA NA NA NA # 7 4.6 3.4 1.4 0.3 setosa NA NA NA NA # 8 5 3.4 1.5 0.2 setosa NA NA NA NA # 9 4.4 2.9 1.4 0.2 setosa NA NA NA NA # 10 4.9 3.1 1.5 0.1 setosa NA NA NA NA
対応策
内部的にどういう挙動になっているかよくわからないが、mutate_atの関数部分は関数か、funsのようにlistを指定する。
そのため、引数付きの関数を事前に作成して関数部分にfunsのようなlistを適用するとよさそう。
partialという関数を用いると既存関数を用いた無名関数を作成することができる。
そのため、この無名関数をfunsのように作成して関数部分に渡すことで対応ができる。
iris %>% group_by(Species) %>% mutate_at(vars(Sepal.Length:Petal.Width), list(lag2 = partial(lag, n=2))) # Sepal.Length Sepal.Width Petal.Length Petal.Width Species Sepal.Length_la… Sepal.Width_lag2 Petal.Length_la… # <dbl> <dbl> <dbl> <dbl> <fct> <dbl> <dbl> <dbl> # 1 5.1 3.5 1.4 0.2 setosa NA NA NA # 2 4.9 3 1.4 0.2 setosa NA NA NA # 3 4.7 3.2 1.3 0.2 setosa 5.1 3.5 1.4 # 4 4.6 3.1 1.5 0.2 setosa 4.9 3 1.4 # 5 5 3.6 1.4 0.2 setosa 4.7 3.2 1.3 # 6 5.4 3.9 1.7 0.4 setosa 4.6 3.1 1.5 # 7 4.6 3.4 1.4 0.3 setosa 5 3.6 1.4 # 8 5 3.4 1.5 0.2 setosa 5.4 3.9 1.7 # 9 4.4 2.9 1.4 0.2 setosa 4.6 3.4 1.4 # 10 4.9 3.1 1.5 0.1 setosa 5 3.4 1.5
なお、mutate_atは前述のようにlistでhoge = 関数で渡すと適用した列の末尾にlistの要素名が_で付けることができるので上記コードもlistで渡しているが、必要ない場合そのままpartial無名関数を渡す。
iris %>% group_by(Species) %>% mutate_at(vars(Sepal.Length:Petal.Width), partial(lag, n=2)) # Sepal.Length Sepal.Width Petal.Length Petal.Width Species # <dbl> <dbl> <dbl> <dbl> <fct> # 1 NA NA NA NA setosa # 2 NA NA NA NA setosa # 3 5.1 3.5 1.4 0.2 setosa # 4 4.9 3 1.4 0.2 setosa # 5 4.7 3.2 1.3 0.2 setosa # 6 4.6 3.1 1.5 0.2 setosa # 7 5 3.6 1.4 0.2 setosa # 8 5.4 3.9 1.7 0.4 setosa # 9 4.6 3.4 1.4 0.3 setosa # 10 5 3.4 1.5 0.2 setosa
モデルに対して値の推定結果を作成する
やりたいこと
モデルを作成して、そのモデルをある値に適用したときの推定結果を作成したい。
今回は
- モデルの学習データの推定値
- 任意の値をモデルに適用したときの推定値
の2パターンについて書く。
共通処理
今回、モデルはdiamondsに対して線形モデルlog(price) ~ clarity + log(carat)を適用した結果で考える。
モデルの学習データの推定値
この場合、modelの結果をbroom::argumentに適用することでmodelに使用したデータおよび、その推定結果が返ってくる。
diamonds %>% do(fit = lm(log(price) ~ clarity + log(carat), data = .)) %>% augment(fit)

このとき、モデルlog(price) ~ clarity + log(carat)に用いた実際の学習データはインスタンス毎にそれぞれそのままlog.price., clarity, log.carat.列として表示される。そしてそれら値を用いた各インスタンスのclarity + log(carat)の結果、つまりlog(price)の推定結果は .fitted列として格納される。また、.se.fitなどで推定結果の統計値も表示される。
また、過去記事のようにモデルをpurrr::mapとbroom::tidy,broom::glanceで作成するのと同じ流れでbroom::argumentも使うことができる。
diamonds %>% group_nest(clarity) %>% mutate(model = map(data, ~lm(log(price) ~ log(carat), data = .)), tidied = map(model, tidy), glanced = map(model, glance), augmented = map(model, augment))

任意の値をモデルに適用したときの推定値
先程は学習データをモデルに適用した結果だが、今度は任意の値を適用した結果を考える。
これはなぜおこなうかというと、 このモデルを適用したときどういう値が推定されるか がモチベーションとなっている。
まずは、学習させたmodelオブジェクトを作成する。
# modelオブジェクトの作成 lm_model = diamonds %>% lm(log(price) ~ clarity + log(carat), data = .)

次に、各clarityに対して任意のcaratとなるインスタンスをdata_gridで作成する。
# 各clarityに対して、任意のcaratとなるインスタンスを作成 dummy_diamonds = diamonds %>% data_grid(clarity, carat = seq(0, 5, 0.01))
これは、今回の場合diamondsのclarityのユニークな値に対して任意のcaratを全て紐付けたDFを作成している。

次に、predictを用いてこのデータをモデルに適用する
# 各clarity・任意のcaratインスタンスを先程作成したmodelに適用した結果を返す dummy_diamonds %>% mutate(fit = predict(lm_model, newdata = .))

可視化
前述のように、 このモデルを適用したときどういう値が推定されるか がわかったので、結果を可視化する。
diamonds %>% ggplot(aes(carat, log(price))) + geom_point(color = 'grey') + # 元のデータポイント geom_line(aes(y = fit), data = pred_diamond, color = 'red') + # 推定された値(fitting curve) facet_wrap(. ~ clarity)

その他(個人的メモに近いので、わかりづらい話)
今回、dummy_diamondsとして推定結果のもととなるデータを作成した。
推定するモデルlog(price) ~ clarity + log(carat) のclarityはカテゴリカル変数だったが仮にintだった場合実際のclarityよりも1.2倍大きかった場合、今のモデルを適用するとどのような結果になったか、という推定結果も出すことができる。
dummy_diamonds = diamonds %>% mutate(clarity = clarity * 1.2) %>% # 1.2倍の値を作成 data_grid(clarity, carat = seq(0, 5, 0.01))
参考
purrrを使って、関数の引数を変えて適用したDFを結合していく
やりたいこと
関数の引数を変えて適用したDFを結合したい。
以下のように、指定した列colに対してunder_value以下となるdfを抽出して、そのunder_valueをfiltered_values列に追加する関数filtered_under_valueで考える。なお、見た目上わかりやすいためheadで2行のみ抜いてきている。
library(tidyverse) data("diamonds") # under_value以下のcolを抽出 filtered_under_value = function(df, col, under_value) { col = enquo(col) df_filtered = df %>% filter(!!col <= under_value)%>% mutate(filtered_values = under_value) %>% head(2) return(df_filtered) } filtered_under_value(diamonds, price, 350)

このunder_valueを変えたdfを結合するには3つの方法がある。
ひたすらコピペする
以下のように、dfを結合するbind_rowsをひたすらコピペして貼り付けていく。
filtered_under_value(diamonds, price, 350) %>% bind_rows(filtered_under_value(diamonds, price, 400)) %>% bind_rows(filtered_under_value(diamonds, price, 450))

for文で回す
コピペはできるだけ避けた方が保守など色々な面で良いので、空のdfに対してforで回したdfを結合していく。
values = c(350, 400, 450) df = data.frame() for (v in values) { tmp_df = filtered_under_value(diamonds, price, v) df = df %>% bind_rows(tmp_df) }

reduceとmapを組み合わせる
forのようなiterateはpurrr::mapで置き換えることができる。こちらの方がシンプルなので可読性が上がる。また、上述の空dfに結合していく方法は空dfを作成する部分を走らせ損なうと値が2重で入るなど、ミスを生みやすい。
df = reduce( map(c(350, 400, 450), ~ filtered_under_value(diamonds, price, .)), bind_rows )

mapとreduceを使っていて、それぞれちゃんと理解していないと難しいのでそれぞれ解説する。
map
map(.x, .f, ...)関数は .xのlistの中身を1つずつ.fに適用してlistで返す関数となる(list以外で返す関数もそれぞれ用意されている)。
つまり、前述のコードのmap部分では以下のようにlistc(350, 400, 450)の3つの値がfiltered_under_value(diamonds, price, .)の.部分に前から入っていき結果をそれぞれlistとして格納する。
m_list = map(c(350, 400, 450), ~ filtered_under_value(diamonds, price, .))
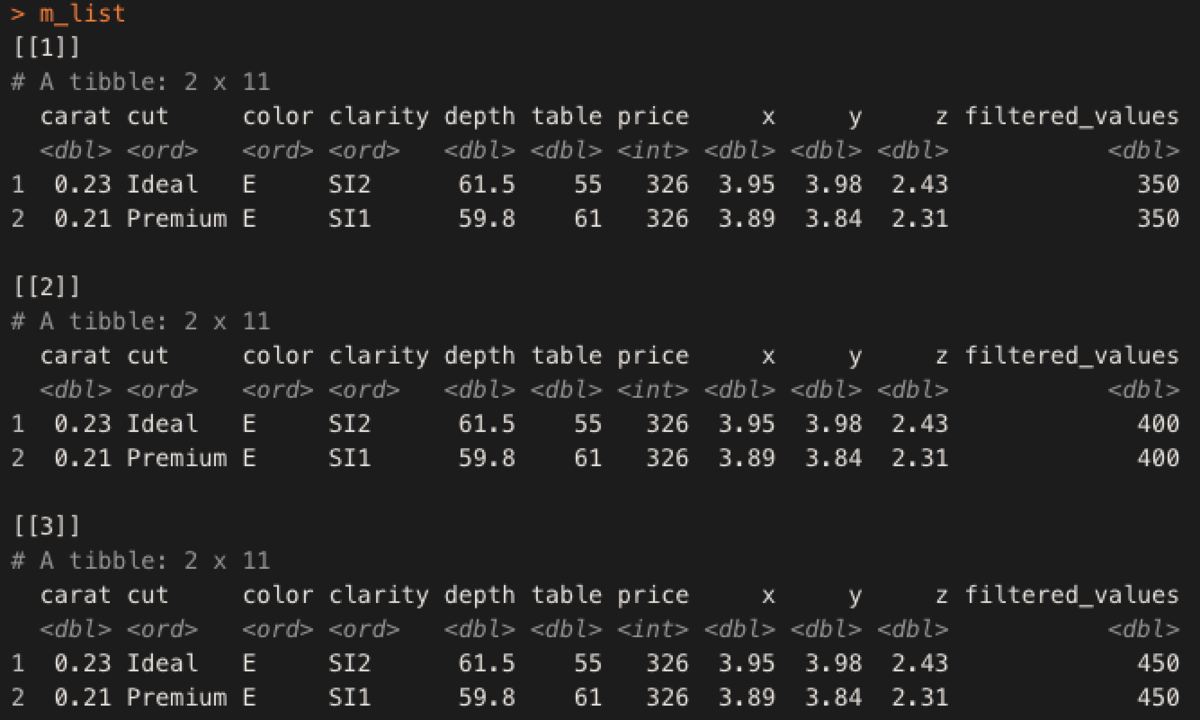
reduce
reduce(.x, .f, ...)は、.xのlistの中身を前から適用し、自分と1つ前の結果を2変数関数(引数を2つ取る関数).fに適用して最終的な結果を返す。
「自分と1つ前の結果を2変数関数(引数を2つ取る関数).fに適用」とは、例えば下記のようなコードの場合、c(1, 2, 3)の1回目は1つ前が無いため.fである+の結果は1、2回目は2と1回目の結果を.fに適用して1+2、3回目は3と2回目の結果1+2(3)の結果を.fに適用して(1+2)+3となり、最後の3回目の結果(1+2)+3を返す。
reduce(c(1, 2, 3), `+`) # => 6
なお、余談だがこの過程をまとめてvectorで返すaccumurateという関数もある。
accumulate(1:3, `+`) # => [1] 1 3 6
今までの説明を踏まえてわかりやすく書き直すと以下のようになる。
m_list = map(c(350, 400, 450), ~ filtered_under_value(diamonds, price, .)) df = reduce(m_list, bind_rows)
つまり、mapでc(350, 400, 450)を順にfiltered_under_valueに適用した結果がlist m_listとして格納され、reduceでlistm_listを2変数関数bind_rowsを用いて、map内でのloopの1回目はlist m_listの1つ目の結果のみ、2回目はlist m_listの2つ目と1回目の結果をbind_rowsした結果、3回目はlist m_listの3つ目と2回目の結果とbind_rowsしてこの3回めの結果を返すことになる。
追記
Each argument can either be a data frame, a list that could be a data frame, or a list of data frames.
bind_rowsはdfのlistを受け取った場合、list内を縦結合するため前述のコードはreduceを使わないでも問題ないようです。
# old df = reduce( map(c(350, 400, 450), ~ filtered_under_value(diamonds, price, .)), bind_rows ) # new df = map(c(350, 400, 450), ~ filtered_under_value(diamonds, price, .)) %>% bind_rows()
参考
ggplotで他の変数を基準に並び替える
数値で並び替える場合
以下のようなグラフで考える。
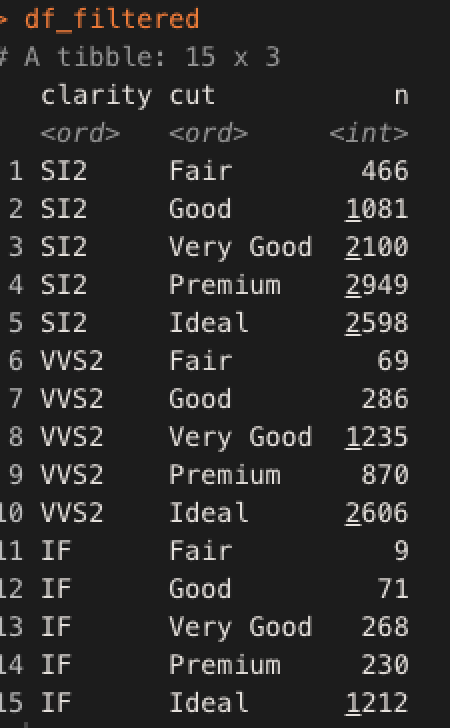
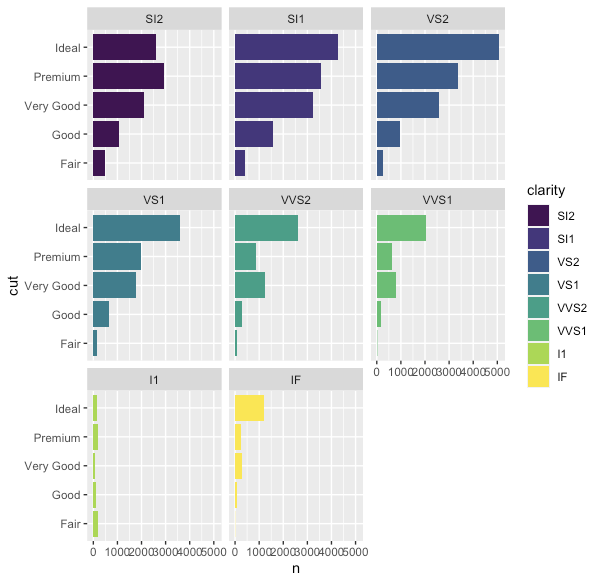
library(tidyverse) data(diamonds) # データ作成 df = diamonds %>% group_by(clarity, cut) %>% summarise(n = n()) %>% ungroup() # わかりやすいものだけ抽出 df_filtered = df %>% filter(clarity %in% c('SI2', 'VVS2', 'IF')) # clarityの標準の順番 df_filtered %>% ggplot(aes(n, cut, fill = clarity)) + geom_col() + facet_wrap(. ~ clarity)
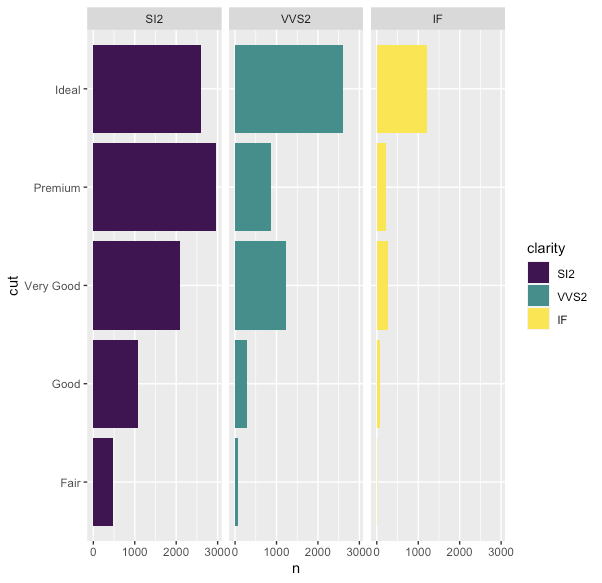
このとき、各facetでの棒グラフはデータフレームでcutが出てきた順で、グラフの下からFair,Good,...といった表示になっている。
このとき、各facet毎に、棒グラフの長さ順で並び替えたい。
factor変数に対して、他の変数を基準に並び替えたい場合は一般的にfct_reorderで並び替える。今回は2変数を基準にするのでfct_reorder2を使う。
df_filtered %>% mutate(cut = fct_reorder2(cut, n, clarity)) %>% # nで並び替える ggplot(aes(n, cut, fill = clarity)) + geom_col() + facet_wrap(. ~ clarity)
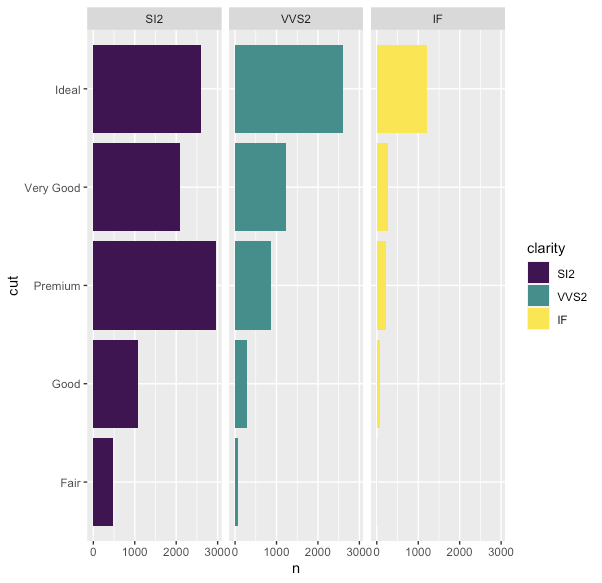
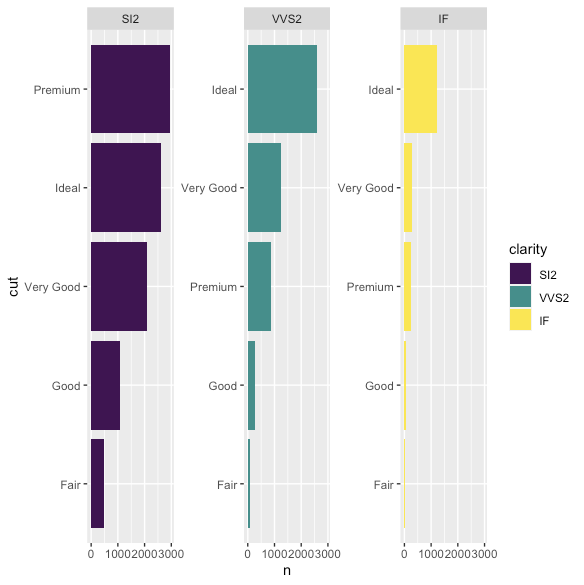
結果を見ると、並び替えは起きているようだが上手くいっていない。
データを出力してみると、以下のようになっており先程と同様にデータフレームでcutが出てきた順で、グラフの下からIdeal,Fair,...といった表示になっている。
df_filtered %>% mutate(cut = fct_reorder2(cut, n, clarity)) %>% # nで並び替える arrange(cut)
グラフで使う場合はreorder_withinを使う。
df_filtered %>% mutate(cut = reorder_within(cut, n, clarity)) %>% # n, clarityで並び替える ggplot(aes(n, cut, fill = clarity)) + geom_col() + scale_y_reordered() + # __で結合されているのを削除 facet_wrap(. ~ clarity, scales = 'free_y') # 自分のfacet変数のみ
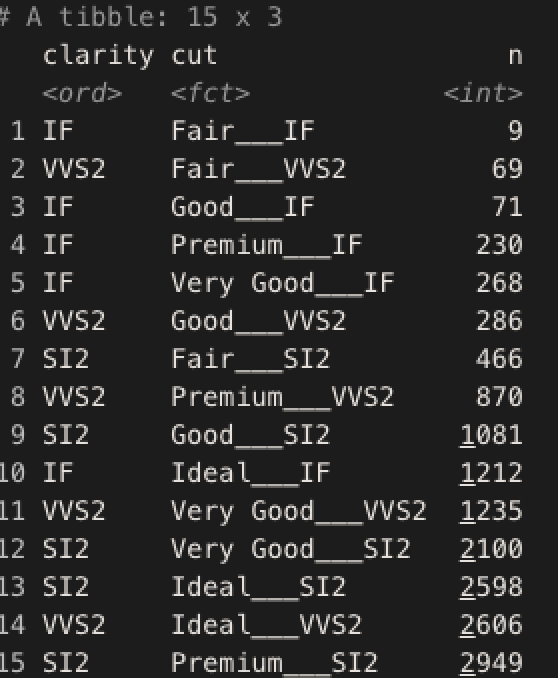
これは内部的に指定した変数(cut, clarity)を結合した新たな変数を用いてnで並び替えをおこなっている。
そのため、新たな結合された変数cut__clarityの出た順になるため各clarityのfacetでは自分のclarityが入っているcut__clarity以外はデータがないので「出た順に並び替える」ことができる。
df_filtered %>% mutate(cut = reorder_within(cut, n, clarity)) %>% arrange(cut)
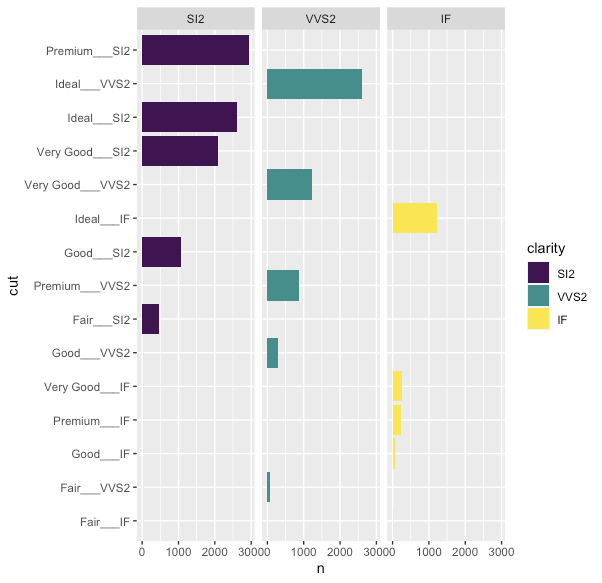
なお、コード中にscale_y_reorderedおよび、scales = 'free_y'を追加しているが、前者は「表示を結合状態から元に戻す」働きがある。
また、後者はデータがない部分を非表示にする働きがある。今回の場合、cut__clarityが自facet内のclarity以外はデータがないが表示自体はされるためscales = 'free_y'で非表示にできる。
これらを外して表示すると意味がわかりやすい。
df_filtered %>% mutate(cut = reorder_within(cut, n, clarity)) %>% # n, clarityで並び替える ggplot(aes(n, cut, fill = clarity)) + geom_col() + # scale_y_reordered() + # __で結合されているのを削除 facet_wrap(. ~ clarity) #, scales = 'free_y')
facetを並び替える
facetは一般的にfactorとなっている。これを別変数を基準にしてfacetを並び替える。
データとして、clarity毎に1文字目を基準にgroupを作り、このgroup毎にclarityのfacetを並び替えて先程と同様のグラフを作成する。
df2 = diamonds %>% mutate(clarity_group = substr(clarity, 1, 1)) %>% group_by(clarity_group, clarity, cut) %>% summarise(n = n()) %>% ungroup() df2 %>% distinct(clarity_group, clarity) %>% arrange(clarity_group, clarity)
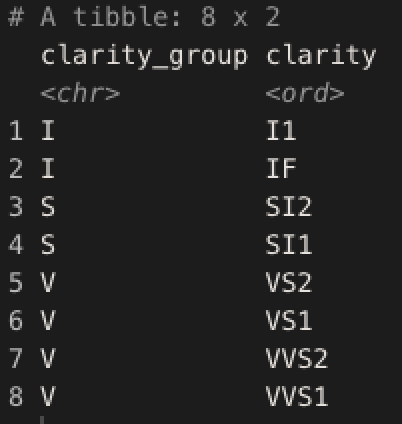
これをそのまま出力すると、当たり前だがclarityの順でfacetが表示される。
df2 %>% ggplot(aes(n, cut, fill = clarity)) + geom_col() + facet_wrap(. ~ clarity)
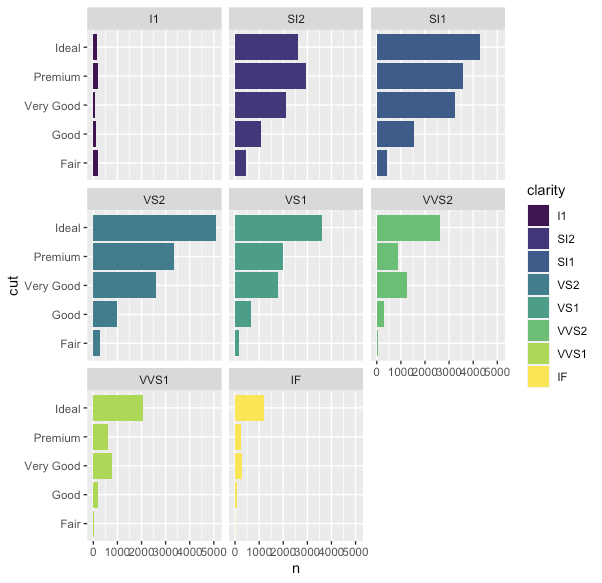
これを同じclarity_groupを固めて表示したい。
facetに2軸を指定する方法
1つ目の方法として、clarity_groupもfacetに表示する方法が考えられる。
df2 %>% ggplot(aes(n, cut, fill = clarity)) + geom_col() + facet_wrap(. ~ clarity_group + clarity)
目的通りclarity_groupが固まって表示されているが、facetと凡例の順が一致していないし、表示があまりきれいではない。
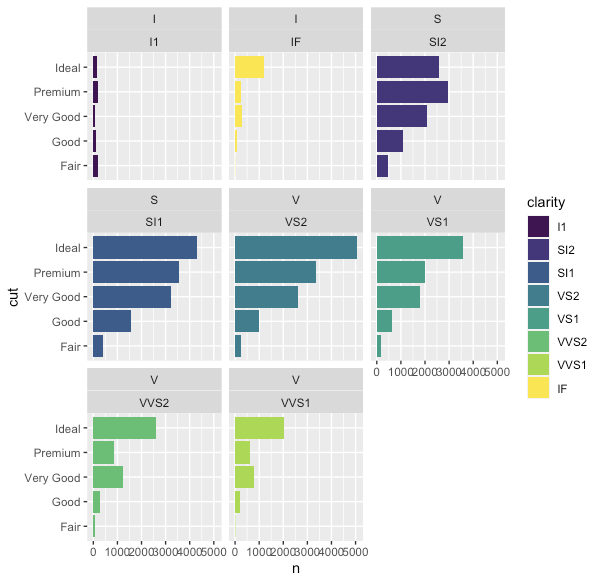
fct_reorderを使う方法
前節でちらっと出たfct_reorderを使い、clarityをclarity_group基準に並び替える。
df2 %>% mutate(clarity = fct_reorder(clarity, clarity_group)) %>% ggplot(aes(n, cut, fill = clarity)) + geom_col() + facet_wrap(. ~ clarity)
この場合は、facetと凡例の順番が一致している。
任意の順にする
また、今回clarity_groupはcharなのでアルファベット順だが任意のclarity_group順にしたい場合は、factorに変換する。
この際、そのままfct_reorderに指定すると型エラーになるのでas.numericでnumericに変換する必要がある。なお、factorをnumericにするとlevel部分の数値となる。
df2 %>% mutate(clarity_group = factor(clarity_group, levels = c('S', 'V', 'I')), clarity = fct_reorder(clarity, as.numeric(clarity_group))) %>% # numericにする ggplot(aes(n, cut, fill = clarity)) + geom_col() + facet_wrap(. ~ clarity)
map2を使ってサブサンプルに分けた各データに対して複数モデルを適用する
表題通り、サブサンプルに分けた各データに対して複数モデルを適用したい。
より具体的に書くと、以下のdropoutさんの記事では
複数のモデルを当てはめる場合
diamondデータに対して、3モデルを適用サブサンプルに分けて分析する場合
diamondデータをclarity毎にサブサンプルをnestで作成しそれぞれに対して、1モデルを適用
といったことをnestとmapを使ってモダンに処理する方法の紹介をしている。
これらを複合して、「diamondデータをclarity毎にサブサンプルを作成しそれぞれに対して、3モデルを適用」といったことをおこないたい。
複数のモデルを当てはめる場合 サブサンプルに分けて分析する場合
これら処理だけをまとめると
library(tidyverse) library(tidymodels) df = diamonds %>% mutate_if(is.ordered, factor, ordered = FALSE) # 複数model formulas = c(log(price) ~ clarity, log(price) ~ clarity + carat, log(price) ~ clarity + log(carat)) %>% enframe("model_no", "formula") # 1. 1データに対して複数モデルを適応 formulas %>% mutate(model = map(formula, lm, data = df)) # 2. サブサンプルごとに1モデルを適応 # dfをclarityごとにサブサンプルに分ける df_nested = df %>% group_by(clarity) %>% nest() %>% arrange(clarity) df_nested %>% mutate(model = map(data, ~lm(log(price) ~ log(carat), data = .)))
となる。
ちなみに、上記記事の各関数詳細などを自分なりに解釈してトレースした過去記事はこちら
このとき、
lmで適用するデータをclarity毎に変えるlmで適用するモデル式を変える
この2つをおこなう。つまり、上記コードの
# 1.formula毎に適用 formulas %>% mutate(model = map(formula, lm, data = df)) # 2. data毎に適用 df_nested %>% mutate(model = map(data, ~lm(log(price) ~ log(carat), data = .)))
このどちらかの部分に対して、formulaかdataを動的に変えることが必要になる。
mapは1引数を2引数目のfunction(モデル式部分)に適用することができる。そのため、formulaとdataどちらとも変えることはできない。
そのため、引数を2つfunctionに渡すことができるmap2を使用する。
なお、はじめのコードにあるモデル式は以下となる。
- log(price) ~ clarity,
- log(price) ~ clarity + carat
- log(price) ~ clarity + log(carat))
今回。clarity毎にしたサブサンプルを使用するため、式の意味を考えると上記3式のclarity部分は必要ない(特に1つ目はlog(price)だけの式になる)ので、今回のモチベーションに合わせるためclarityサブサンプル毎の場合ほぼ同じ意味になる*1以下の2式に修正して適用する。
- log(price) ~ carat
- log(price) ~ log(carat))
実践
モデル式一覧の作成
まずはモデル式一覧を作成する。
# formula候補一覧のデータフレーム formulas = c(log(price) ~ carat, log(price) ~ log(carat)) %>% enframe("model_no", "formula") ## サブサンプルに分けない場合以下 # formulas = c(log(price) ~ clarity + carat, # log(price) ~ clarity + log(carat)) %>% # enframe("model_no", "formula")
モデル式とclarityのセットを作成
次に、map2に指定するために上で作成したformulasに各clarityを付与した列を作成する。
# サブセット一覧の組み合わせ formulas = formulas %>% expand_grid(tibble(subset = unique(df$clarity))) # model_noごとに各clarityを付け加える。6model * 8clarity

後に記述するが、formulasオブジェクトに対してmap2で処理した結果をmutateで加えるため、map2に渡す引数をすべてformulasオブジェクトで完結させるために各clarityを付与した列を作成している。
なお、expand_gridは、適用するオブジェクトに対してexpand_grid内で指定したデータとの全組み合わせを作成する関数となる。
例えば、以下のコードは1,2,3を取るxと1,2を取るyの全組み合わせを出力する。
expand_grid(x = 1:3, y = 1:2)

サブサンプルに分けた各データに対して複数モデルを適用する
# map2() で2変数の組み合わせを並列処理 df_result = formulas %>% # 第2引数.y(subset)で絞り込みをしたデータに対して第1引数.x(formula)を適用 mutate(model = map2(formula, subset, ~lm(.x, data = filter(df, clarity == .y))))
モデルの結果を係数とともに示すと以下のようになる。
df_result %>% mutate(tidied = map(model, tidy)) %>% select(model_no, subset, tidied) %>% unnest() %>% mutate_if(is.double, round, digits=3)

おまけ
元のソースのmapのままforを組み合わせると以下のようになる。
df = diamonds %>% mutate_if(is.ordered, factor, ordered = FALSE) # 複数model formulas = c(log(price) ~ carat, log(price) ~ log(carat)) %>% enframe("model_no", "formula") # clarityの一覧 clarities = df %>% distinct(clarity) %>% pull() # 1. 1データに対して複数モデルを適応をloop df_output = data.frame() # 空のDF for (i_clarity in clarities) { # サブサンプルを作成 df_filtered_clarity = df %>% filter(clarity == i_clarity) df_output_ = formulas %>% mutate(subsumple = i_clarity, model = map(formula, lm, data = df_filtered_clarity)) # df_output_を結合していく df_output = df_output %>% bind_rows(df_output_) }

余談
今回も r-wakalangで質問しました。回答してくださったill_identifiedさんありがとうございました!
参考
*1:正確には、上式ではclarity毎に切片が変わるが他の変数の係数は全clarityで共通。下式では他の変数の係数はclarity毎で変わるため同じではない
ggplotのfacet日本語テキストを折り返す
これはなにか
ggplotのfacet内の文字を任意の文字数で折り返し(改行)をしたい。
どういうことかというと、下記のようにirisベースでテキトーにSpeciesを日本語化して文字数を増やしたもので考える。
library(dplyr) iris2 = iris %>% mutate(Species_jp = case_when(Species == 'setosa' ~ 'セトサ', Species == 'versicolor' ~ 'バージカラー', Species == 'virginica' ~ 'バージニカ'), Species_jp2 = paste0(Species_jp, Species_jp, Species_jp), Species2 = paste(Species, Species, Species, sep = ' ')) %>% tibble()

このSpecies_jp2をfacetにすると文字数が長いので見切れる。
iris2 %>% ggplot() + aes(Sepal.Length, Sepal.Width) + geom_point() + facet_wrap(~ Species_jp2) + theme(strip.text = element_text(family = 'HiraKakuProN-W3'))

これをテキトーな文字数で折り返したい。
このようなモチベーションの際、英語であれば label_wrap_gen を使うことで対応できる。
label_wrap_gen は、 「空白までの間でn文字以上になった場合折り返して、満たない場合は次の空白までをカウントしてその合計がn文字以上なら直前の空白で折り返す」といった処理をおこなってくれる。
例えば「Mostly harmless econometrics」という文章はn = 10であれば、「Mostly harmless (改行) econometrics」として表示される(下記コードはSpeciesを空白付で3回繰り返したもの)。
iris2 %>% ggplot(aes(Sepal.Length, Petal.Length)) + geom_point() + facet_wrap(. ~ Species2, labeller = label_wrap_gen(5))

しかし、日本語文の場合単語を空白で区切らないので label_wrap_gen は使えない。
解決方法
facet_xxxの引数labbelerは指定した関数でテキストを処理してくれる。
そのため、テキストに対してn文字毎に改行コード\nを入れる関数を渡すことでやりたいことが実現できる。
ちなみに、 label_wrap_genも前述のような処理をおこなっている関数というだけ。
# characterの全列に対して、5文字毎に改行を入れた状態に上書きする関数 facet_splitter = function(x) mutate_all(x, ~ str_extract_all(.x, ".{1,5}") %>% map_chr(., ~ paste(.x, collapse = "\n"))) iris2 %>% ggplot() + aes(Sepal.Length, Sepal.Width) + geom_point() + facet_wrap(~ Species_jp2, labeller = facet_splitter) + # 上記関数を適用 theme(strip.text = element_text(family = 'HiraKakuProN-W3'))

ちなみに、labellerを使わないでも先にmutateでfacetに指定する列に同様のことをおこなっておいてもよい。
iris2 %>% mutate(Species_jp2 = (str_extract_all(Species_jp2, '.{1,5}') %>% map_chr(., ~ paste(., collapse = "\n")))) %>% # 直接書き換える ggplot() + aes(Sepal.Length, Sepal.Width) + geom_point() + facet_wrap(~ Species_jp2) + # 上記関数を適用 theme(strip.text = element_text(family = 'HiraKakuProN-W3'))

ただし、決まった文字数で改行しているので単語の中途半端なところで改行は発生している。本当は、単語を識別していい感じのところで改行して欲しいがまぁそれは色々と難しそう(Mecabとか使いながら色々処理する?)なので置いておく。
余談
世の中の情報のほとんどは英語で落ちているため、今回のようなことはググっても日本語では見当たらないし、英語で探しても「 label_wrap_gen を使えばいいよ :)」 という回答ばかりで困っていたので、 r-wakalangで質問した。回答してくださった方々ありがとうございました!
リーダブルコード俺俺メモ①第一部 表面上の改善
最近コードを量産することが多いので、リーダブルコードを読む。
コードは今まで我流だったし、コードを書く機会がそこまで多くなかったので会社でレビューを受けた回数も少ないため一回ちゃんとしないとなぁというのがモチベーション。
自己認識としては、変数名がやばい(後で読み返したとき、自分でも一瞬なんだっけこれ?ってなる)。
完全に俺俺メモなので、各章毎に個人的にできてないことや意識しておくことのみをメモ書き程度の書き方で羅列する。適宜自分なりの解釈や、記事などから引用した内容も書くためリーダブルコード自体には書いてないこと(注釈で「自己解釈」と書いている行)も入るので注意。
また、プログラマーではないので、あまり使わない部分もあるため節によっては飛ばしている。

リーダブルコード ―より良いコードを書くためのシンプルで実践的なテクニック (Theory in practice)
- 作者:Dustin Boswell,Trevor Foucher
- 発売日: 2012/06/23
- メディア: 単行本(ソフトカバー)
1章. 理解しやすいコード
読みやすさの基本定理
コードは他の人が最短時間で理解できるように書かなければいけない。「他の人」というのは少し時間が経ったときの自分も含める。 例えば、そのコードを別のときに流用することもあるし、何かしらの修正が発生したときに改めて読む必要があるが、そのときにできるだけ思い出す時間をかけないためにも必要となる。 また、「理解できる」というのは、他の人が変更を加えたりバグを見つけることができるレベルを指す。
2章.名前に情報を詰め込む
変数名や関数名などの「名前」は短いコメントだと考える。つまり、良い名前は多くの情報を伝えることができるので名前に情報を詰め込む必要がある。
明確な単語を選ぶ
抽象的な単語は避ける。
例えば、getは何を取得してくるかわからないが、fetchやdownloadなら何かしらのデータを外部から取ってくるニュアンスが伝わる。
他には、sizeは何のサイズかわからんので、heigh, numbersなどをつける。
また、stopはkillやpouseの方がより細かい停止のニュアンスがわかる。
tmpやretvalなど汎用的な名前を避ける
tmpやhoge、retval(return valueの略)といった特に意味がない単語は避けて、より明確な単語にする。
明確な単語となることで、その部分で何がしたいかが明確になるのでバグなどを見つけやすくなる。
コメントでいいのでは?と思うかもしれないが、多くの場合コメントは宣言時に添えるため宣言時以降で使用する場合はそのコメントを読まない。そのため、明確な名前がついていると宣言時以降でも間違いや勘違いをした使い方を避けることができる*1。
ただし、tmpに関しては例えば変数の入れ替え時など「この変数は他に役割がない(一時的な値の退避など、直後の1回こっきりでしか使わない一時的なもの)」という情報、つまりtmpという単語の意味自体が意味がある場合などは使用してもよい。
# python # bがaより大きい場合、aとbを入れ替える # 変数上書きが発生するので、一時的に値を退避するためにtmpを使う if a < b tmp = a a = b b = a
ただし、そのような機会は少ないしつい甘えた使い方をしそうなのでtmpを使用したときは「本当にtmpでいいか?」と自問しよう*2。
イテレータ
i,j,kなどはそのものがイテレータだという意味になるので使用してもよい。
ただし、例えばi,j,kが同じループ内で入り乱れているときはどのときにiでjでkかを一見して判別するのが難しい。つまり、 iと間違えてjを使っていても気づきづらい。
そのため、例えばmemberを表すのがi、clubを表すのがjならば、member_i, club_i (どちらもiに統一)にするとより良い(記事中では略したmi,ciも勧めているがmってなんだっけ?ってなったりmember motherで頭文字が被ることがあとでわかったときに書き直しが生まれそうなので使わない方がいいと思う)。
本書では書いていないが、例えばmembersリストから中身を取り出すイテレーションのときにiではなくmemberを使っているコードをよく見る。これは、memberがイテレータなのかどうかパッと見でわからないからあまり良くない、ということになる?*3
# python # i, jを使う(△) for i in members for j in clubs print(i) print(j) # sを除外した単語を使う(△) for member in members for club in clubs print(member) print(club) # 明示的なイテレータを使う(◎) for member_i in members for club_i in clubs print(member_i) print(club_i)
抽象的な名前を避ける
例えば、ServerCanStartの場合、サーバーの何かをスタートできるようにする関数ということはわかるが何をStartできるかがよくわからない。
そのため、CanListenOnPortに変えることで「PortをListenできるようにStartする」ことが明示される。
重要な情報を加える
知らせなければ使い方を誤りやすいような場合は、情報に追加する。フォーマットや時間(sec?ms?)、状態(変換前?後?)などは誤りやすい。
例えば、idのフォーマットがhex(16進法)ならばhex_id、時間の単位がms(ミリセコンド)で入っている(別の単位だと勘違いしたときn倍大きい数値になってしまう)ならduration_msとする、など。
ただし、必ず付けるのではなく、間違った場合にバグが起きそうな場合のみ付ける。
名前の長さ
名前はある程度長くてもコード補完があるのであまり問題にならない。
また、単語の省略は人によってわからないことがあるので、一般的なもの以外は省略しない方が無難。例えば、manageをmngは一般的ではないが、stringをstr、documentをdoc、evalutationnをevalは一般的。
命名規則
エンティティ毎で記法を変えることで、エンティティ情報を伝える。例えば、クラス名はキャメルケース(CamelCase)、変数名はスネークケース(snake_case)、定数の変数は全て大文字CONSTANT_NAME、クラスのメンバ変数は接尾に_を入れるなど。このあたりは会社や言語によって変わったりより詳細定義があるので注意。
誤解されない名前
常にその名前が誤解されないか意識する。
例えば、filterは除外されたのか選択されたのかわからないので、前者ならselect後者ならexcludeをつけたほうが明確。
他にも、clip(切り抜き)もremove(削除) or truncate(切り捨て))、 length(長さ)もmax_length(最大の長さ) or min_length(最小の長さ) など。
範囲
条件を指定するときの範囲は未満(<)なのか以下(<=)なのかは統一する。基本的にはmin maxがついた名前とセットで使うと思うので、以上/以下(<=, >=)を使う*4。
また、文脈によっては限界値となるmin maxではなく、包括的な意味のfirst lastやbegin endなどでも良い。なお、starに対応するendは「超えたら終わる」のか、「超える手前で終わる」のか曖昧なので使わない。
bool値
true, falseの意味を明確にしないといけない。
例えば、xxx_flgに対するよくある批判として**「xxxになったときにフラグが立つのか、xxxするため(xxxする必要がある)のフラグ(xxxでないときにフラグが立つ)のかわからんから使うな」というのがある。例えば、start_flgは「スタートしてたら1」「スタートする必要があると1」どちらでも解釈できる。*5。
そのため、is_xxx has_xxx can_xxx should_xxx などをつけると明確になる。
4章. 美しさ
コードを読みやすくするには余白・配置・順序を意識する必要がある。
原則としては以下の3つがある。
- 読み手が慣れているパターンと一貫性のあるレイアウトを使う
- 似ているコードは似ているように見せる
- 関連するコードをまとめてブロックにする
5章. コメントするべきことを知る
コメントの目的は正確には「コードの動作を説明する」ではなく、「書き手の意図を読み手に知らせる」ことである。
コメントをするべきではないこと
コメントは画面を占拠するので、占拠するだけの価値をもたせる。つまり、コードからすぐにわかることはコメントをしない必要がある。ポイントは「すぐに」。
例えば、以下のようなコードはコードを読めばコメントの内容は「すぐに」わかる。
# python # 2番目の`*`以降を全て削除 name = '*'.join(line.split('*')[:2]
コードの処理をコメントしているだけのような「コメントのためのコメント」をしない。例えば、n = xxx.method(a,b)に対するコメントで「オブジェクトxxxに引数a,bを入れたメソッドZを適用してhogeな処理をしてnに格納する」はコードを記述しているだけ。
「価値のあるコメント」としては、「fugaな処理(処理内容の意図)をおこなう」「もしaがxxxなら、piyoが起き、違う場合puyoが返る(渡す値の違いによる例示)」のようにするべき。
コメントは不適切な名前の穴埋めではない。つまり、適切な変数名をつけていれば、コメントの多くは不必要となる。
そのため、名前に表せれない注意点や細かい部分などをコメントにする(次に見出しで詳細)。
自分の考えを記録する
優れたコメントは考えを記録するためにある。
そのコードに対して試したこと(最適化など)や、何故それをおこなうか(何故必要か)、足りてない部分(コード未整理や足りない機能など)といったことを書くことで、他の人がそのコードを読むときに理解が進み、無駄なことを考えなくてすむ。
また、定数にコメントをつけることでなぜその値を定数にしているか という、値の背景がわかるため、考慮漏れなどを防ぐことができる。
読み手の立場になって考える
プロジェクトに熟知していない人にもわかるように書く。
質問されそうなことを想像する
何故このコードを書いたか背景がわかりづらい部分は読者が疑問に思う場合が多い。
例えば、別の簡単なやり方がありそうなのに、何故このような方法で行っているかなど
ハマりそうな罠を告知する
このコードを読んで勘違いしそうなこと、コードを間違えて使いそうなこと、使用時に考慮しなければいけないこと はなにか。これらを前もって予告することで、罠を回避することができる。
例えば、処理に時間がかかる部分や、特定条件では使用できない、など。
全体像へのコメント
新しくプロジェクトに参加した人にとって、全体像を理解することが最も難しい。
そのため、そのコードファイルに対して「どのようなことをおこなうためのコードが書いたファイルなのか」という全体像を記述するとファイルの立ち位置への理解が進む*6。
また、これはファイル単位だけでなく関数やClass単位、それぞれの塊単位でも同様に「どのようなことをおこなうための関数なのか」を書くと良い。このあたりは、各有名ライブラリのソースコードを読むと関数やClassへの要約コメントの書き方の勉強になる*7。
6章.コメントは正確で簡潔に
コメントは領域に対する情報の比率が高くなければいけない
曖昧な代名詞を避ける
これ、それ、あれなどの代名詞は指す言葉が曖昧になるし、どれを指すか解釈しなければいけないのでできるだけ使わない。
入出力のコーナーケースに実例を使う
入力(引数)や出力(返り値)がどういうときにどうなるか、実例を交える。