BUSINESS DATA SCIENCE 3章 Regularization① 予測のためのR^2
仕事が落ち着いてきたのでBUSINESS DATA SCIENCEを再開。
データなどは作者のgitにある。
この章はなにか
高次元のモデルを作成するときに、オーバーフィットを避けた良い予測をできるモデル選択(変数選択)において注意すべき点が色々とある。
この節はなにか
モデル選択の前に、そもそも予測性能を見るときは何を指標にしたらいいかという話。通常の (以下R2)ではなく、trainと別のデータに対するR2を見る必要がある。
Out-of-Sample Performance
前章で紹介したdeviance(逸脱度)はあるデータに対して作成したモデルがどれくらいフィットしているかを測る指標。
モデル作成の意図が意思決定や予測のときは一般的なtrainingデータ(IS:In-Sample)に対するdevianceを気にする必要はないが、trainingとは別の新規データ(OOS:Out-Of-Sample)に対するdevianceを測る必要がある(逆に、IS devianceはモデル解釈のときに使用する?)。
このとき、同様にモデルの説明力を表す はOOSに対する R2を気にする必要がある。
データのうちnまでを用いて作成されたモデルの係数
とすると、ISとOOSでのR2に用いるdevianceは以下となる。
ISのR2では、
OOSのR2では、
何故予測の際にISではなくOOSで見るかというと、ISを用いたモデルは「ISでたまたま発生しているノイズや偏り」なども含めて構築される。そのため、IS-R2でモデルの予測性能を評価すると、一般性を失ったオーバーフィット状態での評価をおこなうことになる。一方で、OOS-R2で評価をすると、trainとは別のデータで評価するので「ISでたまたま発生しているノイズや偏り」は評価に対してマイナスになり一般性を持った評価となる。
実データ分析
n=1500, 200次元の半導体データに対して、chipが正しいか間違っているかのバイナリ予測モデルを用いる。
library(tidyverse) SC <- read.csv("semiconductor.csv") ## full model full <- glm(FAIL ~ ., data=SC, family=binomial) # Degrees of Freedom: 1476 Total (i.e. Null); 1276 Residual # Null Deviance: 731.6 # Residual Deviance: 320.3 AIC: 722.3 # R^2 1 - full$deviance/full$null.deviance # 1 - Residual Deviance/Null Deviance # => 0.5621432
全200次元を用いたIS-R2は0.56となった。
検定
200次元の検定結果は以下となる。
## grab p-values pvals <- summary(full)$coef[-1,4] #-1 to drop the intercept ## plot them: it looks like we have some signal here hist(pvals, xlab="p-value", main="", col="lightblue")

1章のFDRあたりの議論で、「p値が一様分布から独立に生成されている」と仮定されるが今回は0付近にスパイクがある。
つまり、この仮定を満たさないなにかしらの理由があると考えられる。
Benjamini-Hochberg(BH)法を用いてFDRをおこなう*1 。
## At 10% FDR, we get 25 `signif' fdr_cut <- function(pvals, q=0.1){ pvals <- sort(pvals[!is.na(pvals)]) N <- length(pvals) k <- rank(pvals, ties.method="min") alpha <- max(pvals[ pvals<= (q*k/(N+1)) ]) plot(pvals, log="xy", xlab="order", main=sprintf("FDR of %g",q), ylab="p-value", bty="n", col=c(8,2)[(pvals<=alpha) + 1], pch=20) lines(1:N, q*(1:N)/(N+1)) return(alpha) } fdr_cut(pvals)
このとき、カットオフ基準下にある有意とされる25個ほどの変数を用いてdevianceを再度計算する。
## Re-run a cut regression using only these 25 ( signif <- which(pvals <= 0.0122) ) # SIG2 SIG17 SIG19 SIG20 SIG22 SIG24 SIG46 SIG47 SIG49 SIG50 SIG52 SIG59 SIG61 # 2 17 19 20 22 24 46 47 49 50 52 59 61 # SIG69 SIG83 SIG102 SIG111 SIG136 SIG142 SIG154 SIG166 SIG178 SIG186 SIG189 SIG200 # 69 83 102 111 136 142 154 166 178 186 189 200 cut <- glm(FAIL ~ ., data=SC[,c("FAIL", names(signif))], family="binomial") 1 - cut$deviance/cut$null.deviance # new in-sample R2 # 0.1811822
結果として、このcut-offモデルのIS-R2は0.18となった。これは、先程のfull modelでのIS-R2の0.56と比べて非常に小さな値となる。これは、IS-R2の特徴として、(有意でないもの含めて)変数を増やせば増やすほどIS-R2は増加することが理由となる。
K-Fold Out-Of-Sample Validation
予測性能を評価するOOS-R2を用いる場合、training(IS)に入れていない未知のデータを用いるためK-Fold Out-Of-Sample Validationをおこなう。これは、機械学習で用いるK-Fold Cross Validationと同じもの。全データからいくつかデータを除いてtrainをし、除いたデータで評価をK回繰り返す。
これを用いて先程のfull modelと、先程のcut-offモデルでのOOS-R2を見ることで各モデルの予測性能を比較してみる。
まず、OOS-R2を計算する関数を作成。
## Out of sample prediction experiment ## first, define the deviance and R2 functions ## pred must be probabilities (0<pred<1) for binomial deviance <- function(y, pred, family=c("gaussian","binomial")){ family <- match.arg(family) if(family=="gaussian"){ return( sum( (y-pred)^2 ) ) }else{ if(is.factor(y)) y <- as.numeric(y)>1 return( -2*sum( y*log(pred) + (1-y)*log(1-pred) ) ) } } ## get null deviance too, and return R2 R2 <- function(y, pred, family=c("gaussian","binomial")){ fam <- match.arg(family) if(fam=="binomial"){ if(is.factor(y)){ y <- as.numeric(y)>1 } } dev <- deviance(y, pred, family=fam) dev0 <- deviance(y, mean(y), family=fam) return(1-dev/dev0) }
そして、K-Fold Out-Of-Sample Validationをおこなう。なお、gitコードにcutvar オブジェクトが無かったので cutvar = c("FAIL", names(signif)) をコードに追加している。
cutvar = c("FAIL", names(signif)) # 無かったので追加 for(k in 1:K){ train <- which(foldid!=k) # train on all but fold `k' ## fit the two regressions rfull <- glm(FAIL~., data=SC, subset=train, family=binomial) rcut <- glm(FAIL~., data=SC[,cutvar], subset=train, family=binomial) ## get predictions: type=response so we have probabilities predfull <- predict(rfull, newdata=SC[-train,], type="response") predcut <- predict(rcut, newdata=SC[-train,], type="response") ## calculate and log R2 OOS$full[k] <- R2(y=SC$FAIL[-train], pred=predfull, family="binomial") OOS$cut[k] <- R2(y=SC$FAIL[-train], pred=predcut, family="binomial") ## print progress cat(k, " ") } ## plot it in plum par(mai=c(.9,.9,.1,.1)) boxplot(OOS, col="plum", ylab="R2", xlab="model", bty="n") ## what are the average OOS R2? colMeans(OOS) # WOW! Full model really sucks. # full cut # -7.40218852 0.06442222
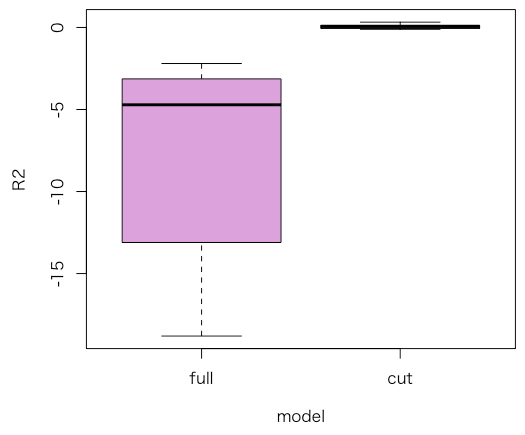
結果として、fullモデルよりcut-offモデルの方がOOS-R2、つまり予測性能は高い。
また、fullモデルはマイナスになっているがこれは、 より、Residual deviance > Null deviance(期待値だけのモデル) となっていることが理由となっている。
なお、今回は比較のためcut-offモデルは固定でおこなったが実際はk-fold毎で変数選択を行う必要がある。
*1:一様分布満たさない場合はBY法では?という疑問が個人的にはあるから理解しきれてない気がする
